Nature Biotechnology |更温和、更准确的DNA/RNA甲基化BS-Seq方法,UBS-Seq
- boke
- 2024-01-05
- 4:03 下午
DNA中的5-甲基胞嘧啶(5mC)是一种重要的表观遗传标记,对于调控基因表达、染色质结构和细胞分类起着关键作用。它存在于各种生物体和不同类型的细胞中,并且是遗传学、表观遗传学和发育生物学等领域中重要研究对象。
亚硫酸盐测序(BS-seq)被视为5mC检测的金标准,其操作简便、经济且检测灵敏度高。但BS-Seq的局限性也非常明显,作为化学转化法,BS-Seq需要极端的理化条件,比如长时间反应和高温,这样会导致严重的DNA损伤。
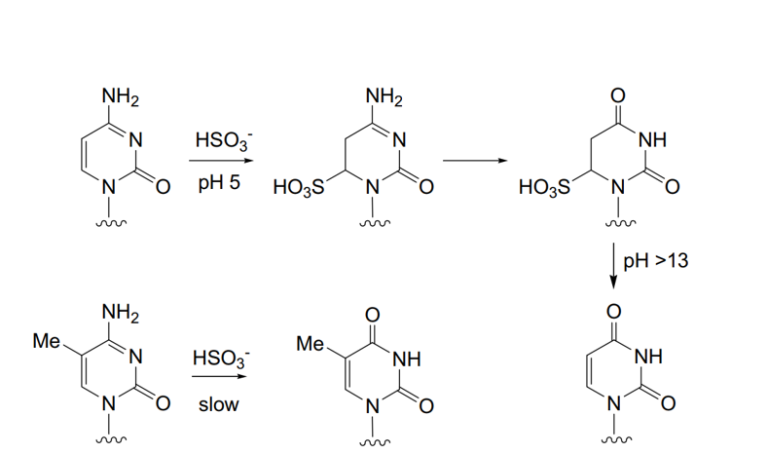
图1. BS转化流程示意图[1]
目前DNA中5mC检测的BS-seq转化流程存在几个限制,具体如下:
(1)在长时间高温反应中,常规BS条件需要在98°C反应10分钟后经过150分钟64°C反应,对于快速检测或诊断应用并不理想;
(2)DNA受到严重损伤,常规 BS处理中的长时间反应会导致大多数经处理的DNA在转化过程中被降解;
(3)C到U的转化不完全,特别是在高GC含量的DNA区域或高度结构化的DNA中;
(4)长时间的BS处理会优先损伤DNA中非甲基化位点,导致甲基化水平被高估。
同样,用于RNA m5C检测的BS-seq也会遇到与DNA类似的问题。RNA的降解以及复杂的二级结构会导致更为严重的降解和不完全转化,造成检测失真。目前,还缺乏一种准确、稳定的RNA m5C检测方法。
最近开发的无亚硫酸氢盐(BS-free)处理的5mC测序方法 ,如酶促甲基化测序(EM-seq)和TET辅助吡啶硼烷测序(TAPS)就是用来解决DNA降解问题(图2),但它们都包括额外的酶处理步骤,这可能会导致其操作复杂度和不稳定性增加。
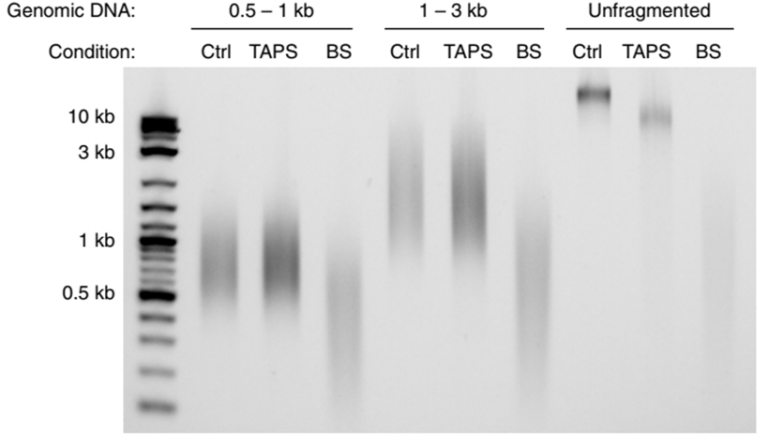
图2. BS-free方法-TAPS可以改善转化过程的DNA降解[2
2024年1月2日,何川教授团队在Nature Biotechnology上在线发表了题为Ultrafast bisulfite sequencing detection of 5-methylcytosine in DNA and RNA的研究论文[3],该研究开发出更准确,损伤更低,适用于低投入量的DNA和RNA甲基化的亚硫酸氢盐测序方法(Ultrafast Bisulfite Sequencing,UBS-Seq)。

难以平衡的两个BS-seq技术参数
BS-seq技术存在两个主要限制,即转化效率和DNA/RNA降解。BS-Seq需要极端的理化条件来保证转化效率,比如高温和剧烈的酸碱性变化,但上述条件会对DNA/RNA造成极大的破坏,而要是采取更温和的处理条件,又会导致转化不完全,形成假阳性,影响检测准确性;所以,保证转化效率或DNA/RNA完整性就像是BS-seq技术参数的跷跷板,难以平衡。
解决方案
根据BS转化机制,BS导致的DNA/RNA降解可能主要与反应时间有关,但反应时间太短,可能转化不完全,那么,问题的重点就变成了如何提高BS的转化速率。高温和高浓度BS是可以提高BS转化速率的(图3),但上述条件对DNA的伤害更大,研究团队设想,如果能短时间完成反应,可能最终会减少降解,并且正好与缩短反应时间的需求吻合。因此,问题被具体为建立一种高效、极速BS转化方法。
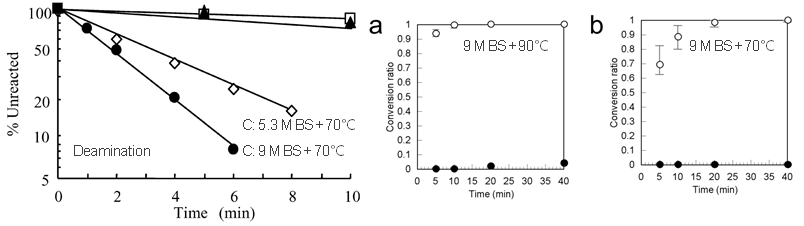
图3. 9M BS试剂或90℃高温可以加速转化过程[4、5]
新型BS配方 USB-1
常规BS由亚硫酸氢钠组成,由于其在水中的溶解度限制,浓度通常在3-5 M。先前的研究发现,铵盐在水中的溶解度更高,采用钠盐和铵盐的混合物可以制备约10M的BS试剂。但这种配方获得的BS试剂亚硫酸盐不易溶解、容易析出,并且粘稠度大,转移困难,在实际使用中可能存在诸多不稳定性隐患,因此,需要寻找一种更优质的BS配方。
转化速率提高13倍:通过大量测试和筛选,研究人员确定了一种新型高浓度BS配方(UBS-1),通过对已知甲基化状态的人工合成寡核苷酸(AGCGA)进行转化处理(98℃)发现,UBS-1可以在3分钟完成对C-U的转化,与传统方法的40分钟相比,转化速率提高了约13倍(图4),同时5mC在10分钟处理内不会出现假阴性。
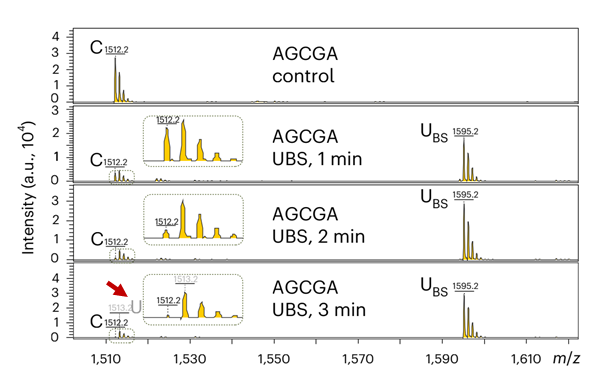
图4. 新型BS试剂配方UBS-1可以在3分钟完成C-U转化
DNA降解减少:将基因组DNA(gDNA)分别在传统的BS和UBS-1条件下处理,凝胶分析清楚地显示UBS-1条件下引起的DNA降解更少(图5,左)。背景噪音降低13倍:为了进一步评估UBS-seq中的假阳性率,对未进行5mC修饰的λ-DNA进行处理,测序结果显示,在UBS-1条件下进行10分钟处理导致C的平均未转化率低至约0.06%,而11分钟处理只稍微降低了背景。相比之下,传统BS处理在λ-DNA上产生了超过13倍(0.82%)的背景(图5,右)。
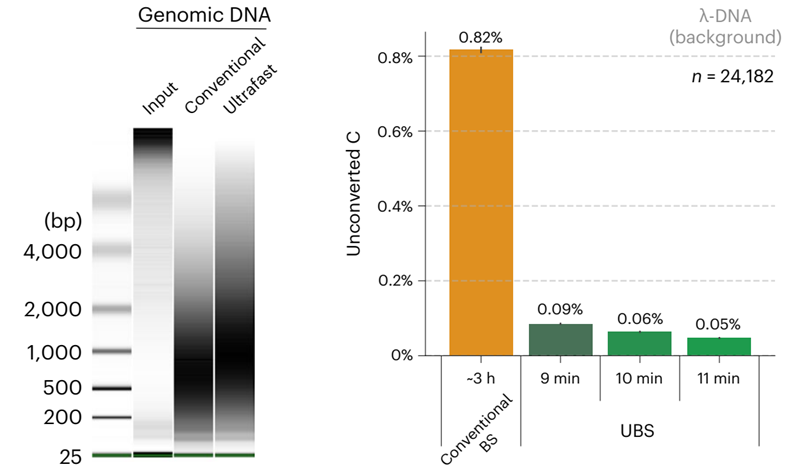
图5. UBS-1配方对DNA损伤更低,假阳性(未转化C)更低[3]
初步性能验证
使用 mESC gDNA 验证 UBS-seq
为了全面比较UBS-seq和传统的BS-seq的性能,对小鼠胚胎干细胞(mESC)的10ng和1ng的基因组DNA进行甲基化建库和测序分析。与上文的结果一致,UBS-seq的转化效率更高。对于CpG位点,两种方法产生了类似的分布模式,传统BS-seq显示出稍高的5mC水平,但对于CHG和CHH位点,传统BS-seq检测到的5mC水平明显高于UBS-seq(图6)。此外,当应用于1ng低投入时,技术重复之间的差异变得更大(图6,右)。
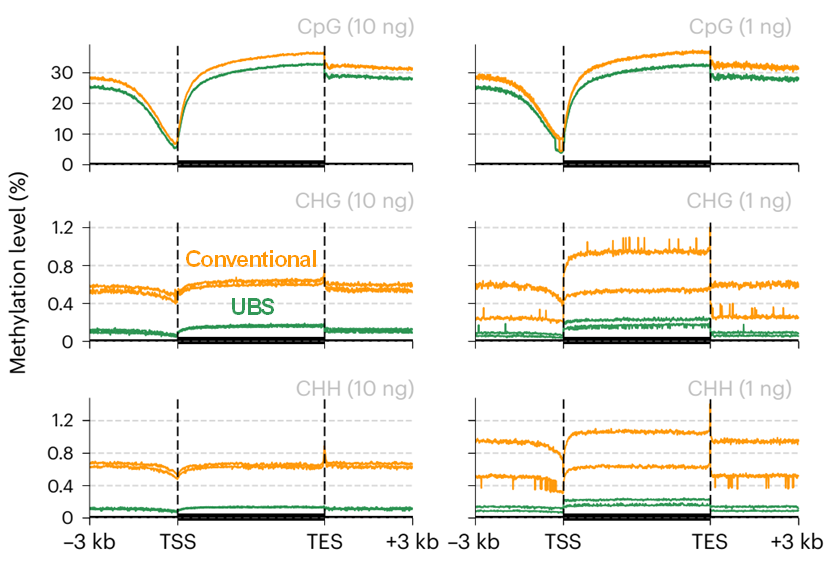
图6. 传统BS-seq可能高估5mC水平[3]
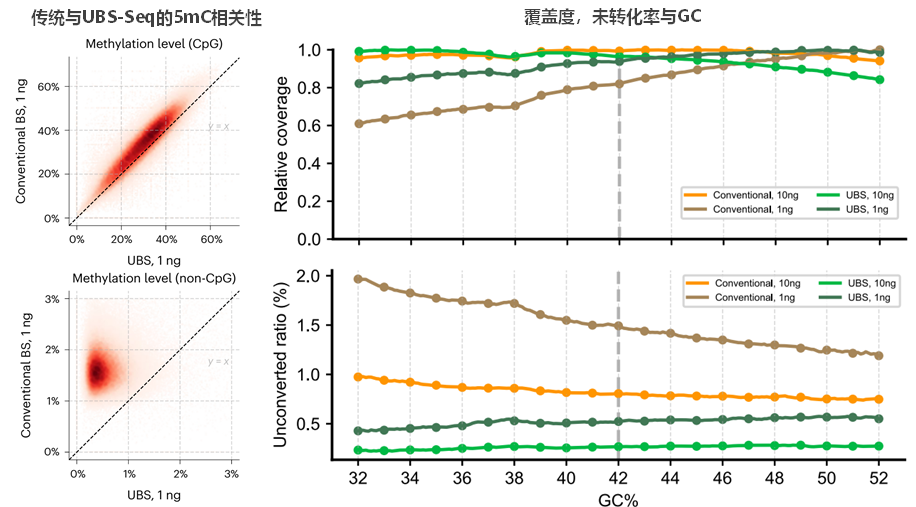
在对10kb区域内的甲基化水平进行比较显示,常规的BS-seq方法系统性高估了5mC水平(图7,左),这表明在常规BS-seq中,低修饰水平的非CpG位点易受到偏好降解的影响,导致DNA中非CpG甲基化水平被高估。研究人员计算了所有1kb基因组区段中的平均测序覆盖度和甲基化水平,发现传统BS-seq方法在低GC区域的覆盖度和转化率系统性减少,而UBS-seq显示出更均匀的分布(图7,右)。总的来看,UBS-seq方法优于传统BS-seq,包括在减少背景干扰、降低5mC水平高估和提高基因组覆盖度方面。
应用于cfDNA甲基化检测
血浆游离DNA(cfDNA)中5mC检测作为癌症诊断、监测和预后生物标志物展现出了巨大潜力。然而,在应用BS-seq技术于cfDNA样本时存在一些挑战:
(1)大多数人体组织中,不到0.5%的基因组区块显示出>50%的差异甲基化,因此需要覆盖率良好的测序方法;
(2)cfDNA来自不同细胞起源的混合物,差异甲基化模式可以区分不同细胞类型,但不同细胞类型的差异在非CpG区域尤为明显,因此非CpG区域的检测准确性非常重要;
(3)患者体内通常只能获得少量cfDNA,因此常规BS-seq中的DNA降解成为一个较大挑战。由于BS试剂导致大部分DNA降解,且未甲基化的DNA片段比甲基化的片段更容易降解,因此甲基化片段往往被富集,导致对总5mC水平的潜在高估(图8)。
相比常规BS-seq,UBS-seq具有更低的背景、更少的DNA降解、较小的偏差和更快的处理时间,因此在cfDNA 5mC测序中具有显著优势。
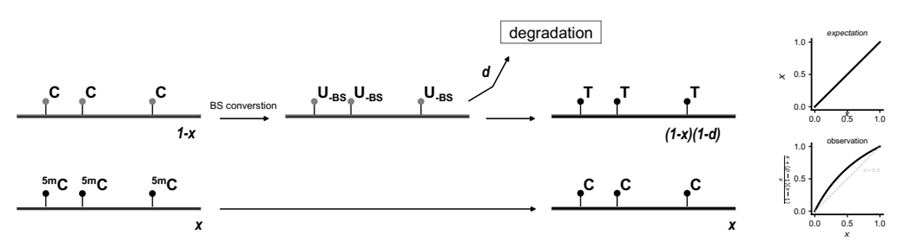
图8. BS降解基因组并导致甲基化被高估的示意图[3]
使用UBS-seq和传统BS-seq技术,以7.5ng人类血浆cfDNA为起始构建文库。在基因组窗口(10kb)内,UBS-seq的两个重复样本和传统BS-seq在所有CpG位点的总测序覆盖度保持高度一致,但是UBS和传统BS处理的样本之间存在轻微差异,表明DNA降解的影响在基因组区域之间并不一致。两种方法在重复样本中检测到的甲基化水平一致性相当高(图9c、d),但传统BS-seq检测到的甲基化水平仍然普遍高于UBS-seq,并且在部分甲基化区域这种差异更为显著(图9e)。
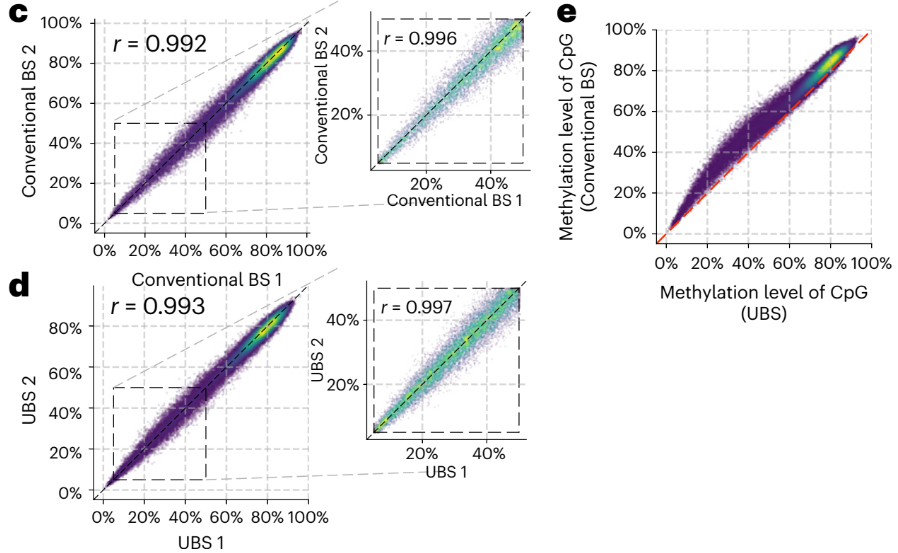
图9. UBS-seq检测cfDNA性能与传统方法高度一致[3]
甲基化面临的一个障碍是不同来源样本的浓度和质量存在相当大的变化。这些变化进一步削弱了检测方法的可靠性。进一步使用10、1和0.1ng的人类血浆cfDNA进行文库构建。无论在技术重复还是不同投入量条件下,UBS-seq一直表现出在基因组内均匀分布。然而,当使用传统的BS-seq时,非CpG位点上检测到的5mC比例变异较大(图10),进一步说明UBS-seq在准确解析cfDNA甲基化模式中的卓越性能。

图10. UBS-seq与BS-seq在不同cfDNA投入量下的甲基化检测表现[3]
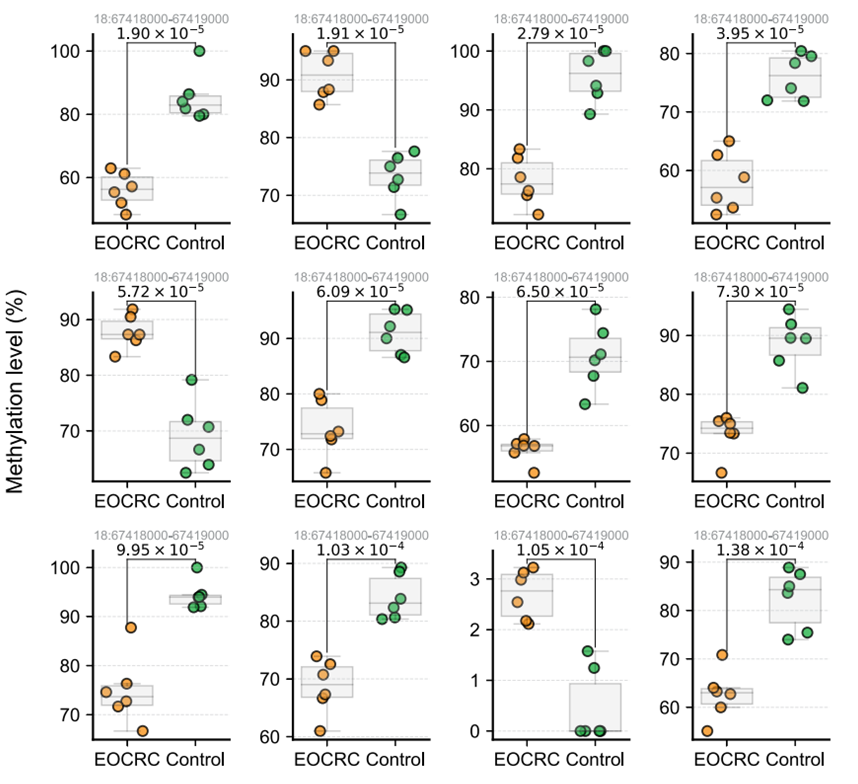
随后,研究人员将UBS-seq应用于早发性结直肠癌(EOCRC)患者和健康对照组的临床样本中,以评估其作为诊断工具的潜力。在这个概念验证研究中,UBS-seq技术成功地确定了135个可能的甲基化区域作为潜在的生物标志物(图11)。
接下来,研究人员还将改良后的UBS-seq应用于rRNA、tRNA和mRNA的甲基化检测分析,并得到了非常优质的数据表现。此外,利用UBS-seq检测到的甲基化位点对NSUN蛋白的功能进行了进一步的探索,证明了UBS-seq在检测RNA甲基化方面的优异性能,将极大地有助于未来对RNA m5C的功能研究。
讨论
在高等真核生物中,gDNA中的5mC是最常见和最重要的表观遗传标记之一。探索其在生物学功能和人类疾病中的作用推动了大量检测DNA甲基化的方法发展。其中,BS-seq仍然是被广泛认可的标准方法,并且已被广泛应用于基础研究和临床应用。尽管取得了一定的成功, BS-seq仍然存在一些局限性。
基于对BS处理介导的C-to-U转化和DNA降解机制的洞察,研究人员通过使用亚硫酸盐的铵盐代替钠盐来解决所有这些局限性,以实现更高的亚硫酸盐浓度和更高的反应温度,从而加速反应并变性DNA/RNA,使BS转化能够在几分钟内完成。新开发的UBS-1试剂不仅提高了BS的转化效率以显著降低背景信号,尤其是在结构复杂的DNA区域,而且减少了DNA降解并减少了5mC水平的过高估计。
UBS-seq仍然存在与传统BS-seq相同的缺点。由C转变为U会导致reads的复杂度降低,可能导致比对问题。不过,可以通过构建具有更长片段的文库并使用成对末端测序来改善比对比例,从而最小化这个问题。并且通过进一步改进数据分析,可以减少由低复杂度引起的比对问题。
综上,新开发的UBS-seq不但适合于少量细胞以及cfDNA中的5mC测序,改良过的UBS-seq方法还解决了RNA m5C测序中的关键技术挑战,UBS-seq为DNA/RNA的甲基化研究提供了更为快速、准确的技术工具。
参考资料
1. Tanaka K, Okamoto A. Degradation of DNA by bisulfite treatment[J]. Bioorganic & medicinal chemistry letters, 2007, 17(7): 1912-1915.
2. Liu, Y., Siejka-Zielińska, P., Velikova, G. et al. Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution. Nat Biotechnol 37, 424–429 (2019).
3. Dai, Q., Ye, C., Irkliyenko, I. et al. Ultrafast bisulfite sequencing detection of 5-methylcytosine in DNA and RNA. Nat Biotechnol (2024).
4. Hayatsu H, Negishi K, Shiraishi M. DNA methylation analysis: speedup of bisulfite-mediated deamination of cytosine in the genomic sequencing procedure[J]. Proceedings of the Japan Academy, Series B, 2004, 80(4): 189-194.;
5. Shiraishi M, Hayatsu H. High-speed conversion of cytosine to uracil in bisulfite genomic sequencing analysis of DNA methylation[J]. DNA research, 2004, 11(6): 409-415.
推荐阅读