分割检测、个体化质控 | 半固定MRD Panel - RaDaR技术浅析
- boke
- 2022-09-15
- 7:21 上午
RaDaR™是一种基于Inivata公司InVision®平台技术的Tumor-informed多重PCR的NGS测序。RaDaR™旨在追踪48个患者特定的变异,用于检测多种肿瘤类型的MRD,可用于治愈性治疗后或复发的早期检测。RaDaR™于2021年3月获得美国食品和药物管理局的突破性设备认定[1]。Inivata公司报告说,RaDaR™的VAF检测限为0.001%,从样本采集到报告的反馈时间约为七天[2]。
RaDaR™技术前身可以追溯到2012年的报道[4],被称为Tagged Amplicon Sequencing (TAm-Seq)。如图1所示,该技术是一种基于PCR原理的靶向测序技术,整体思路和巢式PCR相似,由三步PCR完成。
利用Primer Pool进行多重PCR扩增,得到多靶标混合物;
在上一步产物的基础上进行1-plex PCR,保证每一对引物都高效工作,该步骤通过微流控技术实现;
将上一步产物Pooling后,利用通用引物和index Primer进行标记,随后上机测序。
对于等位基因频率为2%的ctDNA,TAm-Seq可以达到超过97%的灵敏度和特异性。
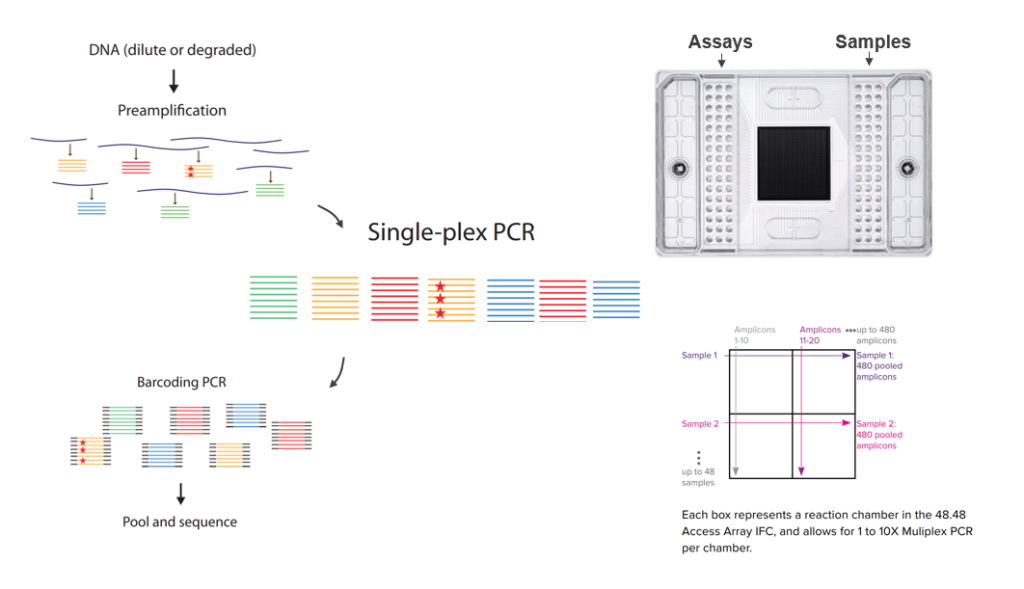
图1 RaDaR™技术前身TAm-Seq工作原理示意图[4-6]
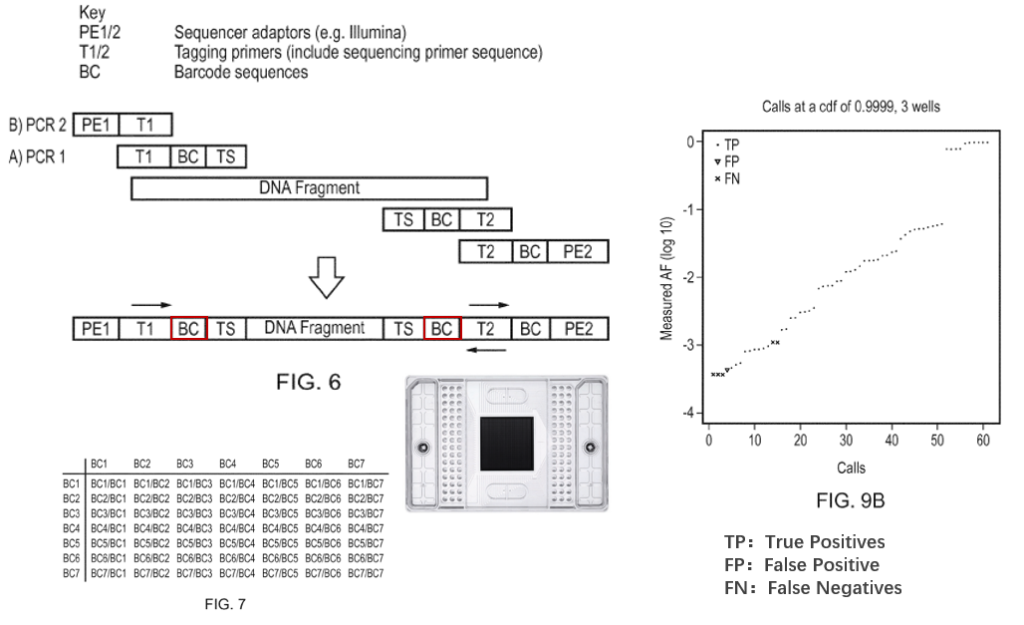
如图2所示,不同于TAm-Seq(“伪多重”,依靠微流控1-plex PCR实现),TAm-Seq技术进一步优化,在不依赖UMI的前提下,采用“分割策略(Partitioning)”,通过对每个反应中的DNA拷贝数、阳性判定条件(≥2孔或3孔)等参数进行调整,提高信噪比[8]。
该流程通过两步PCR完成,1)目标区域多重PCR;2)Index PCR。针对41对扩增引物,为了实现将样本分割成48份,研究人员在每对引物的正向和反向引物的5’端增加了Barcode(BC),用于不同分孔的标记(Inner Barcode),这个位置通常是UMI的区段,整个反应的装配仍然通过微流控工艺完成[8]。
升级后的TAm-Seq(enhanced TAm-Seq™ (eTAm-Seq™))能够检测到低至0.02% AF的突变等位基因,单碱基特异性高达99.9997%。在两个独立实验室的测试中,对于最佳的DNA input样本,eTAm-Seq™对等位基因频率(AF)为0.25%-0.33%的检出率为94%[7]。
在LIONESS研究中(Liquid BIOpsy for MiNimal RESidual DiSease Detection),RaDaR™技术表现亮眼。在手术前采集的血浆样本中,基线ctDNA的VAF水平在0.014%至0.97%之间。在手术后的样本中,可以检测到ctDNA的水平低至0.0006% VAF[3]。
整体而言,RaDaR™技术在TAm-Seq基础上有如下细节调整值得关注: