Tumor-informed Safe-SeqS MRD技术梳理
- boke
- 2022-07-18
- 7:21 上午
在上一篇文章中(NEJM | Tumor-informed Safe-SeqS MRD检测指导II期结肠癌辅助治疗),我们介绍了NEJM近期刊登的一项关于利用血浆ctDNA检测技术指导结肠癌术后辅助化疗的临床研究,试验结果积极[1]。
本文将简单回顾上述研究中ctDNA检测的湿实验环节,并对该研究中使用的ctDNA检测方案-“Safe-SeqS”的技术发展进行梳理。
如图1所示,该研究采用Tumor-Informed方案开展MRD检测,通过15-Gene Panel对患者的FFPE样本进行靶向高通量测序,筛选体细胞突变位点;采集30-60mL血液样本,并从10mL以上的血浆中提取cfDNA,从FFPE的15-Gene Panel基因变异数据中挑选至少一个体细胞突变位点对cfDNA中的ctDNA进行追踪。
ctDNA检测采用Safe-SeqS多重PCR纠错技术,将cfDNA样本分割检测,分别分成12、24或95孔,每孔0.5-3 ng。12和24孔,以及95孔分割的样本采用不同的方法对样本和对照DNA的突变等位基因频率(MAF)差异进行统计分析,P<0.1判定为ctDNA阳性。
Safe-SeqS是什么技术,其经历了哪些优化和调整,而且,为什么要对cfDNA进行分孔检测?此外,Safe-SeqS多重PCR可以一次检测多少位点,引物设计难度如何,是否能够满足MRD的时效性要求,带着这些疑问我们继续往下看。

图1 肿瘤组织突变分析与ctDNA追踪的实验方法
Safe-Sequencing System (“Safe-SeqS”) 在2011年首次提出,其基本思想是通过单分子标记(UID,unique identifier)减小样本制备阶段和测序环节的错误,准确评估低频突变。当然,大家现在对这个思路比较熟悉了。如图2所示,UID标记后的分子再通过PCR并测序,相同UID分子标记的Reads中≥95%的Reads支持的变异被认为是“Supermutant”,这一思路可以在基于PCR或捕获方式的富集路线中实现[2]。

图2 Sefe-SeqS技术UID标记原理示意图[2]
基于PCR靶向扩增的Safe-SeqS技术,最初的实验流程如图3所示,首先使用带有12N或14N随机碱基(UID)的UID Primer进行第一轮PCR,两个循环,使原始Unique模板被标记上不同的UID,同时,也具有上下游扩增的通用序列(UID PCR),再使用Exonuclease I(Exo I)消化残留的UID Primer,然后进行第二轮PCR(25cycles),添加样本区分作用的Index标签(Index PCR),完成文库构建后高通量测序。
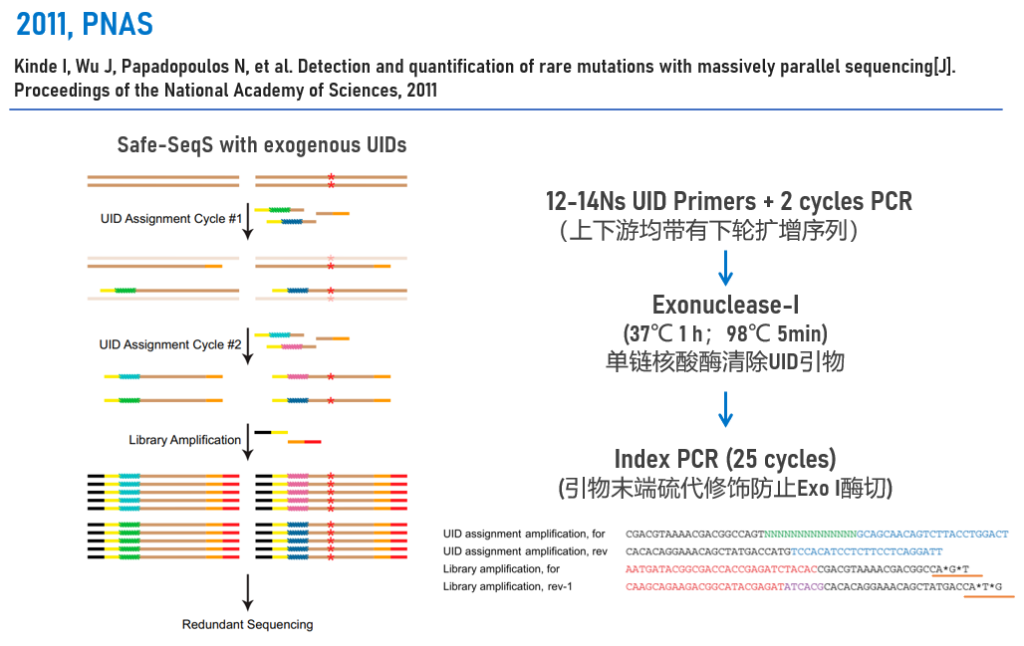
图3 Safe-SeqS第一代实验流程[2]
2013年,Kinde I[3]等在第一代实验流程的基础上对Safe-SeqS进行了优化,Exo I核酸酶被替换成RecJf,用于提高UID Primer清除效率,并在UID PCR后加入纯化环节,进一步减少残留UID Primer的干扰,Index PCR循环数从25减至23(图4)。
此外,该研究也提供了多重PCR引物的设计和优化信息,1)采用Primer 3对12个基因(12-Gene)的感兴趣区域设计多重PCR引物,其中,60%的引物一次性设计成功,可以获得目标条带,剩余的40%需要二次以上设计以提高特异性和扩增效率。2)引物优化后的12-Gene Panel含有46对引物,分为6个反应进行Safe-SeqS检测,每个反应含有6-9对引物。
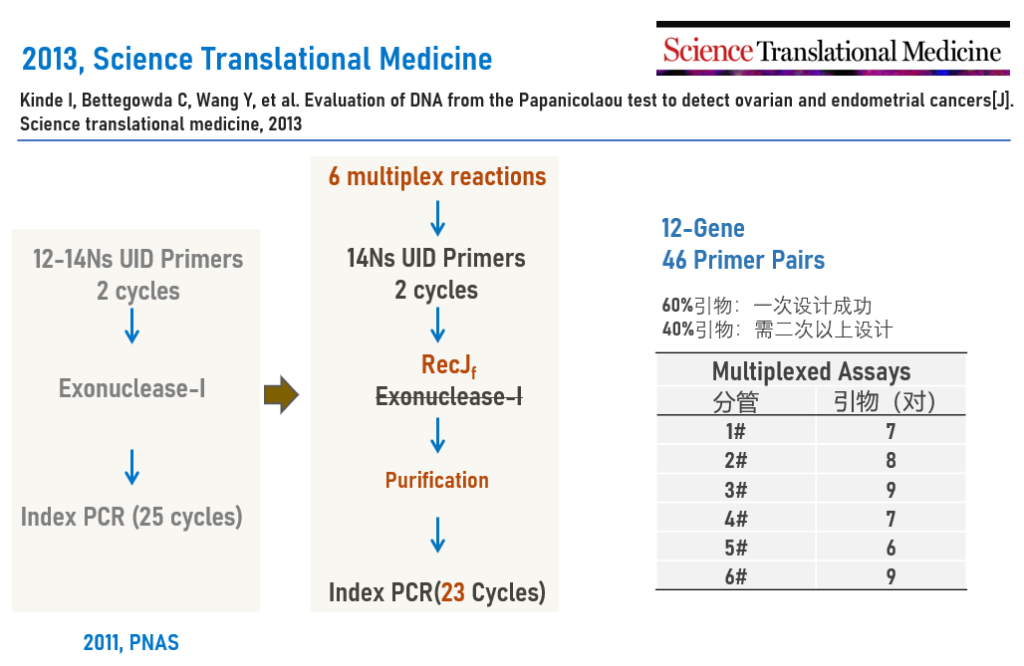
图4 12-Gene Panel的Safe-SeqS技术流程图[3]
2016年,Wang Y等[4]对Safe-SeqS技术进行了较大调整,覆盖17-Gene的133对引物被分成3个Tube进行扩增,UID PCR循环数由最开始的2个循环调整为15个循环,并删除了UID Primer酶切消化环节,Index PCR使用UID PCR产物的1/400(0.25μL)进行扩增(图5)。多重PCR的多重数从6-9重/Tube,提升至~40重,有了明显提升。
可能是由于Safe-SeqS原流程中2轮UID PCR的标记效率有限,为提高模板的利用率(文库转化率),所以增加了UID PCR循环数;上述调整并未放弃UID标签的纠错功能,只是将 “绝对定量”调整为相对定量。
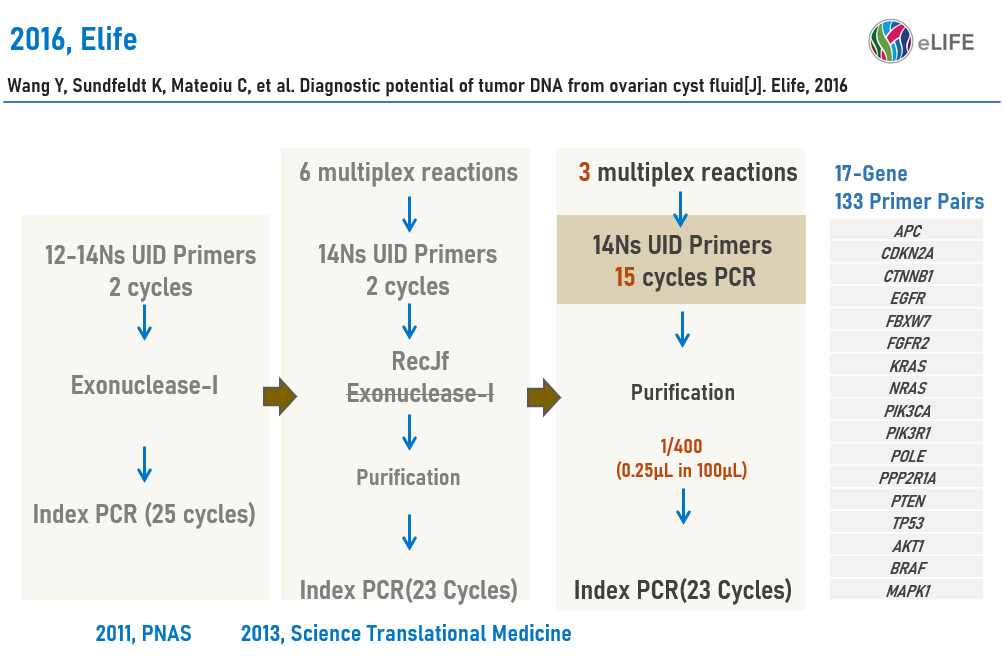
图5 Safe-SeqS技术由“绝对定量”调整为相对定量[4]
“绝对定量”示意图如图6左所示,假设有3个模板,其中1条含有突变位点,MAF(Mutant Allele Frequency)=33.3%,2轮UID PCR后,每个模板被标记上UID(通用序列未显示),分别为UID 1#、UID 2#和UID 3#,Index PCR后每个UID+Template被大量扩增,UID 2#+Template中≥95%的Reads含有相同突变位点,被判定为“Supermutant”,MAF=1(Supermutant)/3(UID Number)=33.3%。
但是对于低频突变,2轮UID PCR的UID标记效率有限,痕量的突变模板丢失的风险较大,因此,增加UID PCR的循环数可以增加模板的标记效率,但是相同的模板会被标记到不同的UID中(图6右),MAF虽然仍是33.3%,但UID的数量与原始模板不再是理论的1:1,即相对定量。
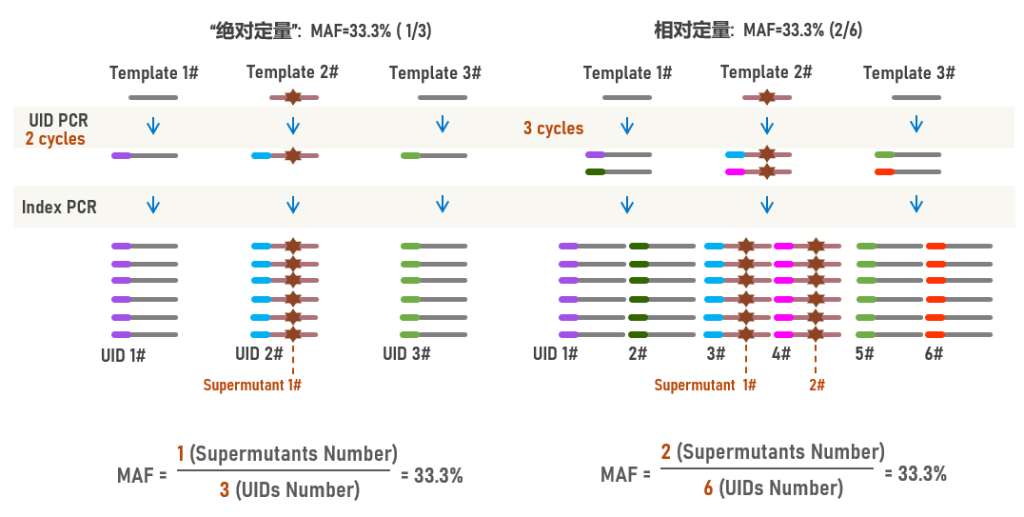
图6 UID PCR循环数调整后,对目标片段的评估由“绝对定量”调整为相对定量
2016年,在“相对定量”Safe-SeqS的基础上,Tie J等人[5]将UID PCR产物用量从0.25μL(1/400, in 100μL)提高至2.5μL(1/100, in 250μL),可能是为了提高取样的代表性以及操作的稳定性。此外,我们看到了对cfDNA的24孔分割操作(0.5-3 ng each),但具体原因未见描述(图7);此外,15-Gene Panel的多重PCR引物的分组情况也未见阐述。小编继续查阅文献寻找答案。
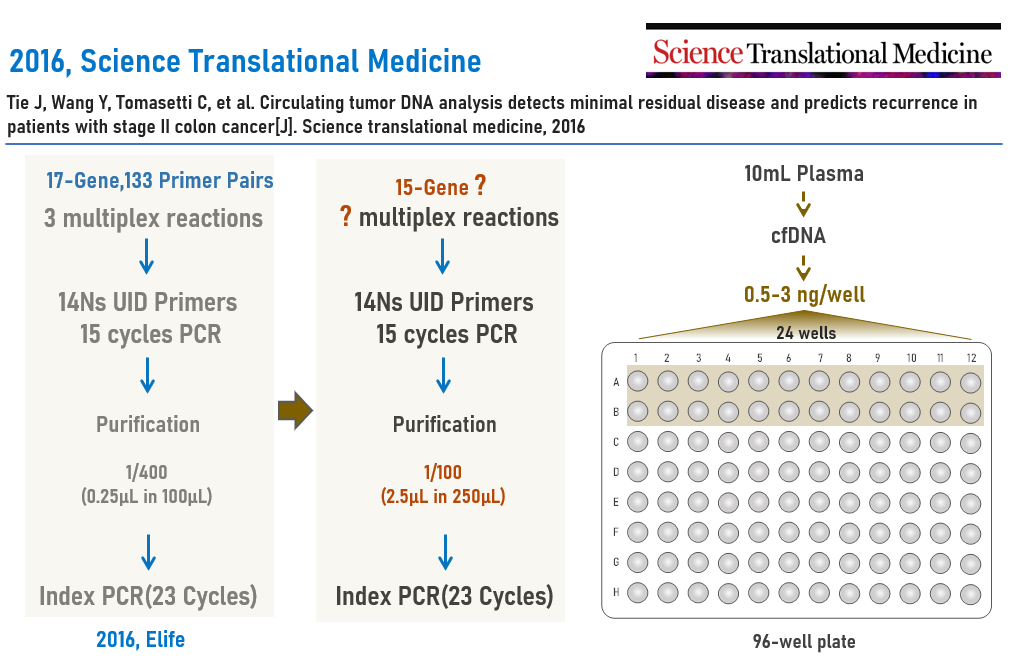
图7 “相对定量”Safe-SeqS的进一步优化[5]
2018年,Science报道了一种名为CancerSEEK的液体活检方法,利用血液ctDNA和蛋白标志物可以对8种癌症进行早期筛查[6];如图8所示,ctDNA检测方法采用Safe-SeqS多重PCR技术,使用15 Cycles的UID PCR(“相对定量”),目标区域为16-Gene,由61对引物组成,分为两个反应进行扩增(28/33对),每个反应包括6个重复,在96孔板中共占一排,便于高通量批量处理,提取的75μL cfDNA按照5μL/孔加入。
研究中对多孔划分方案的原因进行了阐述,由于检测灵敏度通常受到突变等位基因比例的限制,将提取的cfDNA分为多个小份,虽然减少了每个孔的DNA拷贝数,然而,它增加了突变分子的比例(2/600→0/100、0/100、0/100、0/100、1/100、1/100?),使得突变更容易检测。而分割为多少份更优,文章并没有提及。本研究采用的12孔分割,并不是优化设计,而是技术条件限制。其次,分割策略还可以在多个孔中检测到的突变提供的重复信号。因此研究者认为这种分割策略能提高信噪比。
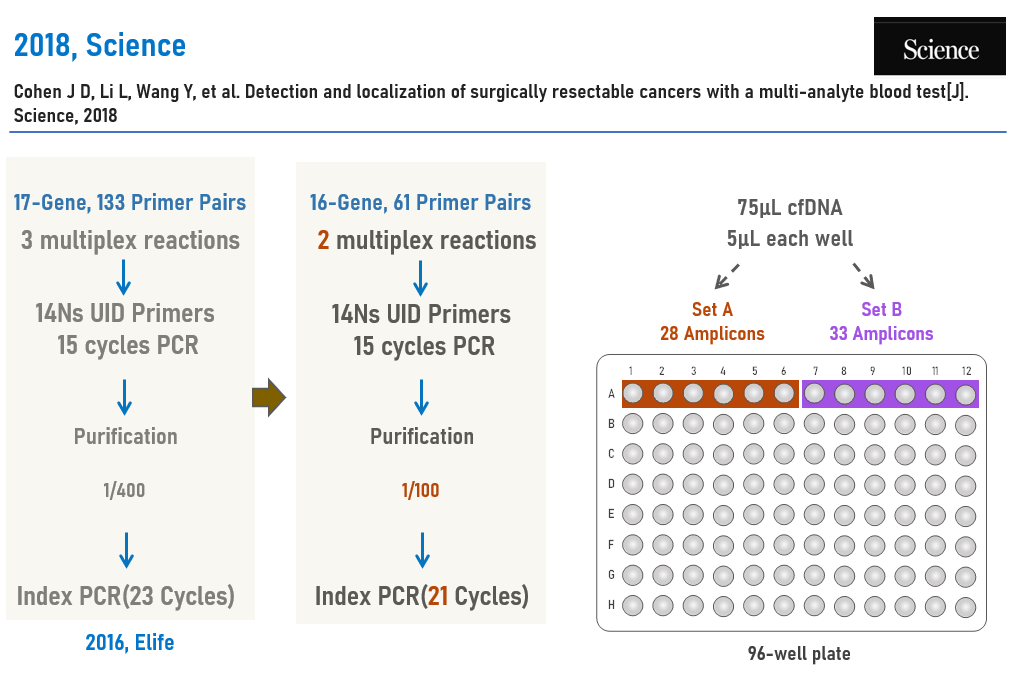
图8 16-Gene Safe-SeqS工作流程图[6]
综上所述,Safe-SeqS多重PCR靶向检测技术经历了十余年的发展,单分子标签(UID)纠错是低频突变检测的核心,UID PCR从2 cycles优化至15 cycles,用于提高模板利用率,UID Primer酶切消化步骤随之删除;cfDNA分割检测有利于提高突变等位基因频率并提供重复信号,提高信噪比。上文报道的多重PCR的重数≤200[6],用于个体化MRD变异追踪比较富裕;PCR引物设计的一次性成功率为60%[3],如果二次或三次设计优化,MRD周期上有一定隐患;此外,不同扩增子(引物对)之间还可能存在相互干扰[4,6]。先前NEJM报道的MRD检测指导II期结肠癌辅助治疗研究采用15-Gene 多重PCR Panel筛选个体化变异追踪位点,不存在全新位点重新设计的问题。那这种策略能否适用于更多的MRD应用场景呢,我们期待更多的深入研究。