奔跑吧,后浪!| 国产WES芯片两大测序平台的测评(上)
2021-05-20
与WGS相比,WES更像是倚天剑,精悍灵活、削铁无声。依靠较少的数据量,WES便可有效覆盖大部分基因组编码区,检测SNP、InDel变异不在话下,此外,还可以灵活添加其他区域,例如线粒体,CNV和病毒模块等,具有极高的性价比。
正因如此,WES芯片已经从科研阶段逐步进入广泛的临床实践,主要包括遗传病诊断和肿瘤诊疗。例如,通过WES构建患者个体的全外显子突变谱,在个性化肿瘤疫苗制备中,筛选更为有效的新生抗原;作为TMB检测金标准,为肿瘤免疫治疗选择提供依据;为MRD检测提供突变信息,让变异标志物的选择更加游刃有余,为肿瘤治疗方案评估、复发监控等提供更多参考。作为提前兑现肿瘤监测的MRD检测,基于全外突变谱的检测方法更具针对性,肿瘤异质性与现阶段普遍应用的肿瘤Panel相对固定的基因存在一定矛盾,很难” One Size Fits All”(图1A、B),同时,由于ctDNA检测敏感性与检测位点数量正相关(图1C、D),因此,使用全外突变谱可以间接提高MRD检测准确性。
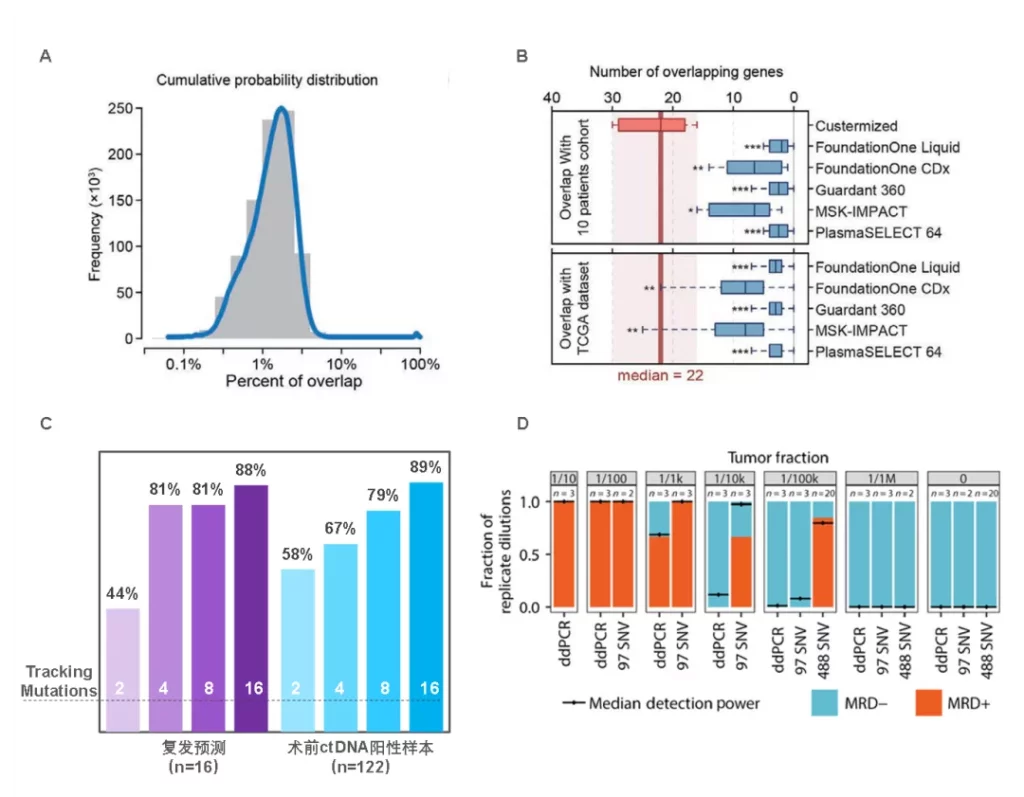
图1. A. 50例NSCLS患者体细胞突变重合比例(TCGA)[1]。B. WES芯片检测到的突变基因与商品化Panel基因List的重合基因数量。上方Panel,10例NSCLC队列数据;下方Panel,1059例NSCLC TCGA数据;红色方框为根据WES突变谱设计的个性化Panel,蓝色方框是固定基因的商品化Panel[1]。追踪不同数量突变的ctDNA检测性能评估(C,D)[2,3]。
总而言之,不论是TMB还是MRD检测,更加全面的全外显子突变谱将更好,由于体细胞突变检测的测序深度要求较高(≥500x),测序成本需要进一步降低促进临床应用的推广。WES芯片作为全外显子突变图谱构建的统一标准也将是水到渠成,众望所归。
WES芯片由几十万条探针组成、设计与合成技术均需要大量的实验数据积累,例如,核苷酸的选择,RNA还是DNA,单链还是双链?一段180bp的目标区域,使用120nt探针头尾相连好(240nt Bait Region),还是60nt OverLap更好(180nt Bait Region),两种设计Reads覆盖特征有何区别?而这仅仅是在设计层面,因此,想要生产一款高性能WES芯片并非易事。
作为靶向捕获技术的开拓者,Agilent、Roche和IDT在过去很长一段时间引领着靶向测序前进的方向,不论是特异性还是覆盖均一性也都是行业标杆。现在,国产WES芯片作为后起之秀在性能参数方面和国际竞品相当,甚至更好。
今天,小编就为大家带来一款国产WES芯片(TargetCap Core Exome Panel v3.0)在Illumina和MGI双测序平台的测评表现,该WES芯片由伯科生物自主研发生产,由近40万条120nt ssDNA探针组成,作为WES界的“后浪”,其表现究竟如何呢?话不多说,上图。
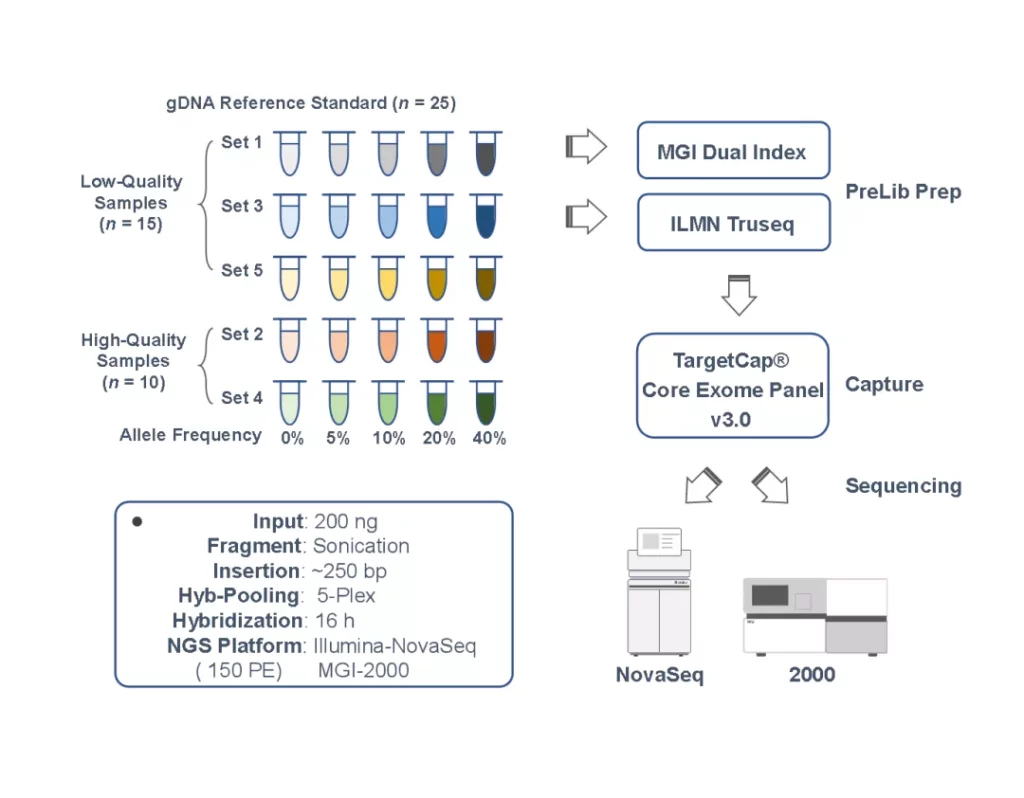
图2. TargetCap®Core Exome Panel v3.0双平台测试实验流程
首先介绍整个实验设计,考虑到WES在全外突变谱建立中的应用,样本方面,采用含有不同突变数量的5套gDNA标准品(Set 1-5),每套标准品由不同等位基因频率的5个样本组成(0%-40%),共25例样本,按照样本质量的高低分为两类,分别为稍低质量样本(LQ,15例)和高质量样本(HQ,10例)。对上述25例样本分别构建Illumina Truseq和MGI 双端Index文库,按照5-Plex与TargetCap®Core Exome Panel v3.0杂交16h,分别在Illumina NovaSeq(ILMN)和MGISEQ-2000(MGI)平台进行150PE测序50Gb以上,随机抽取10Gb进行数据质控。
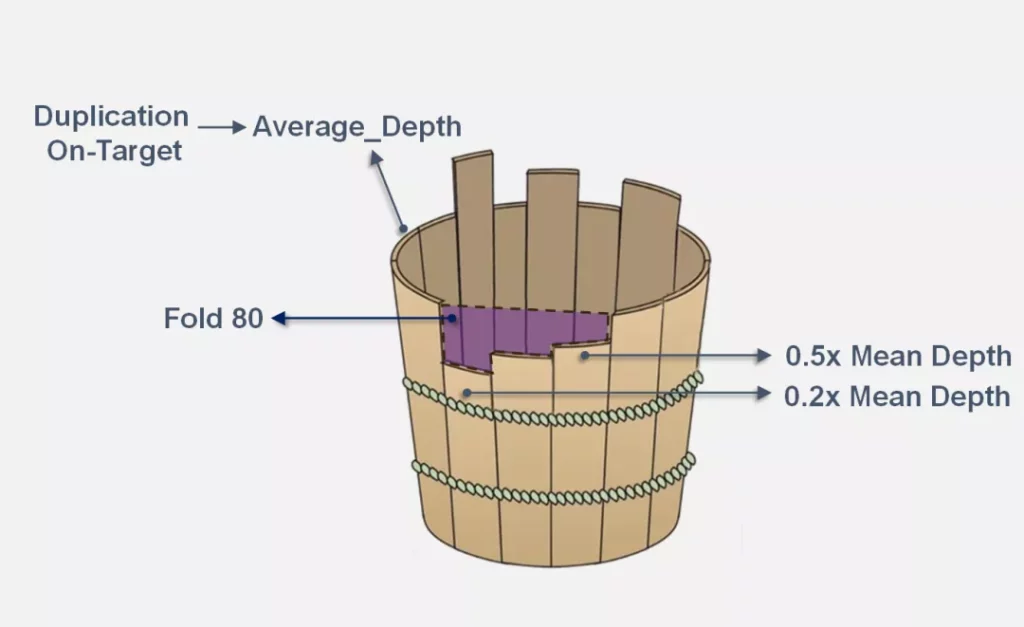
图3. 靶向测序评估参数示意图
对于靶向测序数据的评估,冗余度和中靶率决定了有效数据,即平均测序深度,而覆盖均一性则决定了有多少目标区域能够用于最终的分析,FDA所批准的全外产品Omics Core规定平均测序深度≥500x,0.2x Mean Depth≥95%(≥100x的目标区域占比);和木桶效应一样,木桶够高还不行(平均测序深度),能盛多少水取决于最矮的木板(0.2x Mean Depth);除了最矮的木板,还有一些木板稍高,他们就是0.3、0.4、0.5…x Mean Depth等,对于体细胞突变分析,当然是“多多益善”,即更多的区域达到更深的测序深度,因此0.3-1.0x Mean Depth的值越高越好。除此之外,还有一个评估均一性的参数叫Fold 80,计算公式为平均深度/80%以上目标区域覆盖的深度,低于2.0均一性较好,可以理解成评估整个缺口的大小。
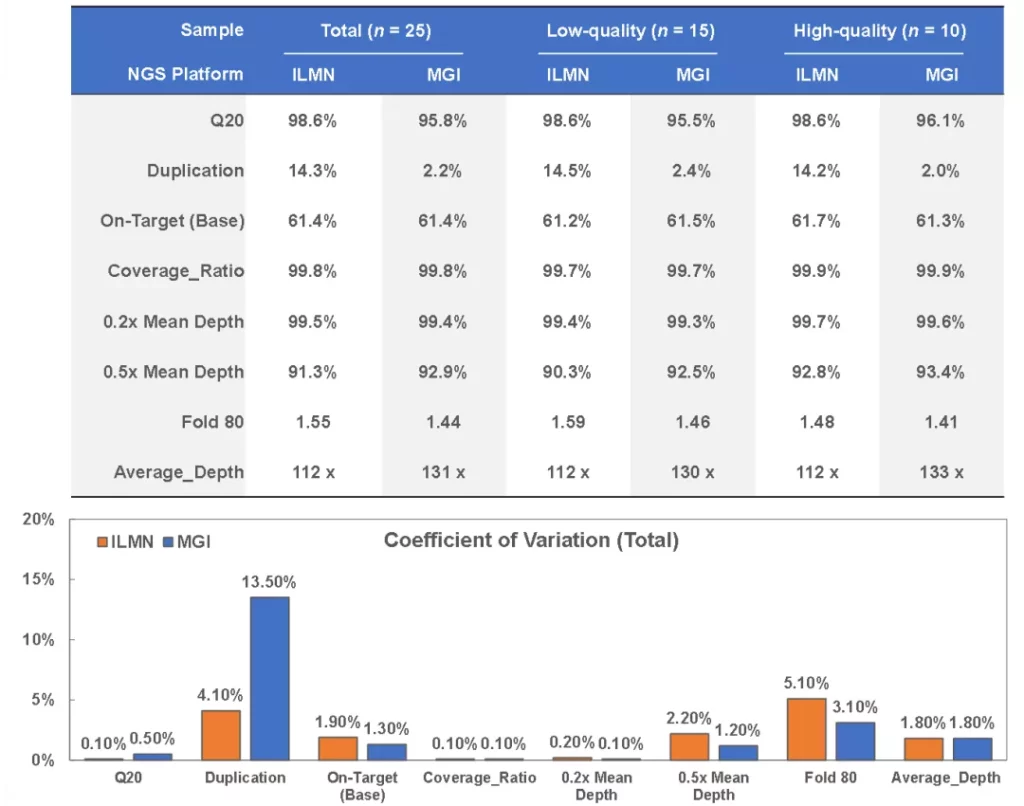
图4. 伯科WES芯片双平台测序参数
碱基质量
ILMN平台碱基质量Q20在不同质量样本中保持一致,均为98.6%,MGI平台在总样本中为95.8%,LQ和HQ样本分别95.5%和96.1%。在碱基质量上出现样本质量相关的趋势。
数据有效性(冗余度+中靶率)
中靶率(On-Target)方面,两个测序平台无明显的差异。在冗余度方面,MGI平台:~2%,ILMN平台:~14%。
目标区域覆盖率
ILMN与MGI测序平台完全一致,达到总体99.8%,LQ 99.7%,HQ 99.9%,表现出高质量样本的覆盖率稍高于较低质量的样本。
覆盖均一性
1.低深度区域占比
0.2x Mean Depth: ILMN与MGI测序平台几乎一致(Total:99.5%/99.4%),在总体、LQ和HQ样本中,两个测序平台数据表现均较为优异。
0.5x Mean Depth: MGI/ILMN(92.9%/91.3%)两个测序平台在不同质量样本中趋势一致。
2.整体均一性(Fold 80)
MGI(1.44)与ILMN(1.55)两个测序平台在不同质量样本中趋势一致。
接下来,我们再看一看TargetCap®Core Exome Panel v3.0的稳定性,对于总体25例样本,除Duplication参数外,不论是ILMN还是MGI测序平台,其他各项参数的CV值均控制在5.1%及以下,数据表现出极佳的稳定性;其中,MGI平台Duplicaiton的平均值为2.2%,CV值为13.5%,相对较高的CV与极低的均值有关。
TargetCap®Core Exome Panel v3.0分别在两个测序平台上的数据表现令人印象深刻,覆盖率:99.8%,中靶率(碱基):61.2-61.7%,0.2x Mean Depth:99.3-99.7%,0.5x Mean Depth:90.3-93.4%,Fold 80:1.41-1.59。对比伯科个性化定制Panel,Core Exome保持了伯科Panel的基本表现(图5)。
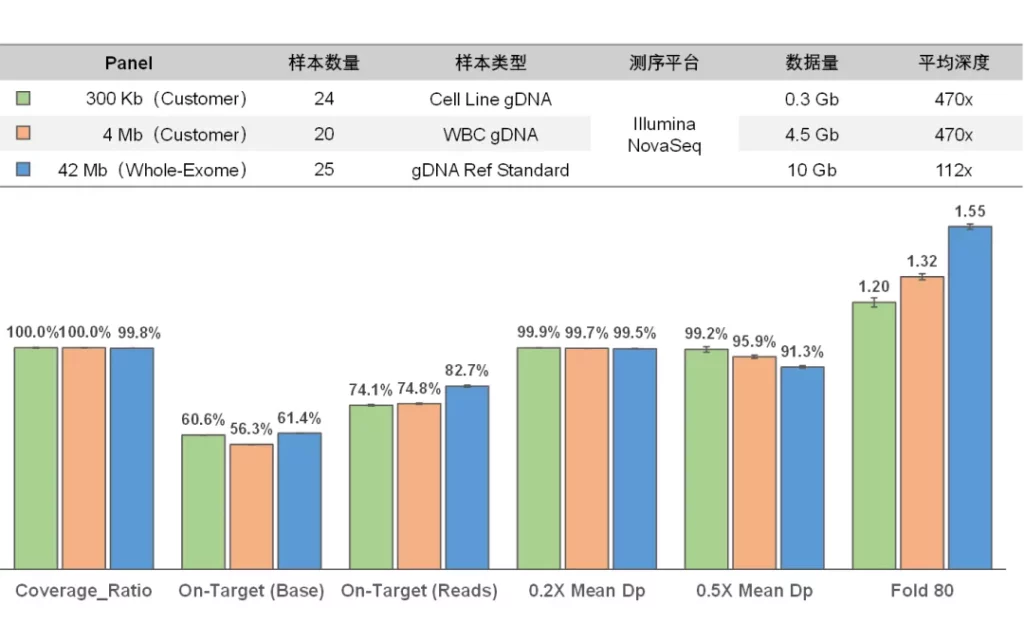
图5. 伯科个性化Panel数据表现
ILMN和MGI平台差异比较
MGI和ILMN测序平台在测试中展现出来的差异究其背后的技术原因,可能是来自于信号放大过程中(文库扩增),两个测序平台选择的路线差异,如图6所示,MGI采用线性滚环扩增,ILMN使用指数桥式扩增,在冗余度及整体均一性上,MGI体现出线性扩增的特征。
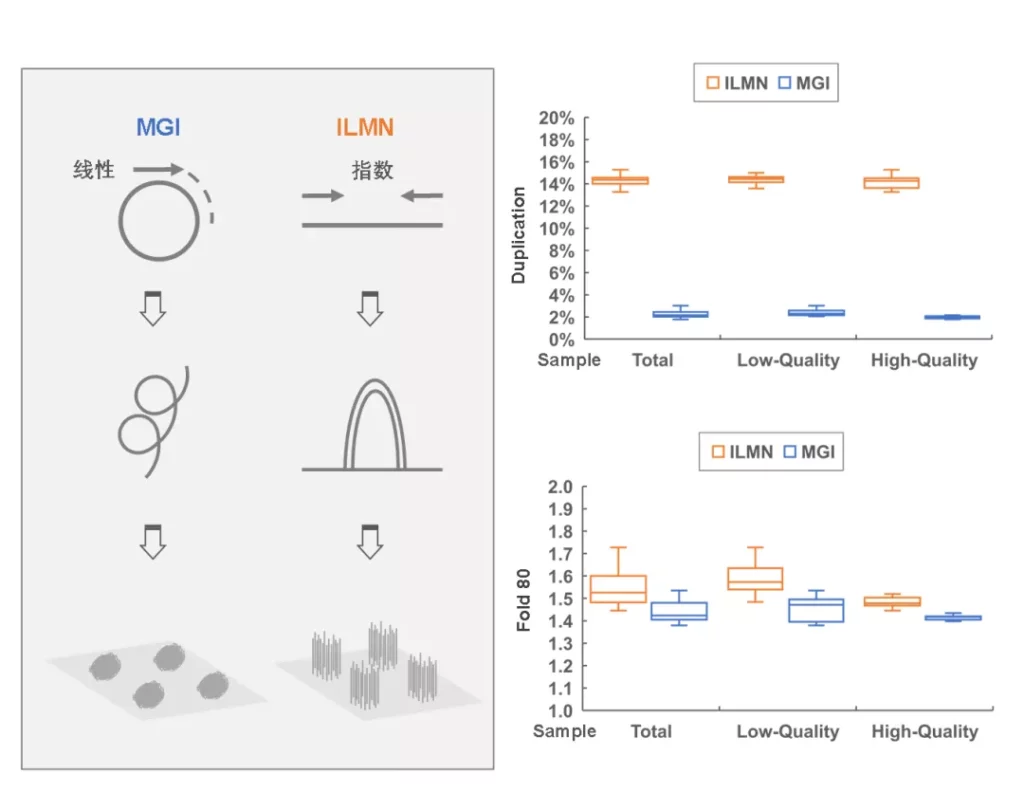
图6. ILMN与MGI信号放大示意图
0.2x Mean Depth
作为关键的“最矮木板”,0.2x Mean Depth在两个测序平台上几乎完全一致(ILMN 99.5%,MGI 99.4%),但进一步分析低于0.2x Mean Depth(下文简称低深度区域)的区域(Bait)发现,低深度区域的GC分布存在明显的平台差异,在高质量样本中,ILMN平台低深度区域的GC分布相对均匀,在低GC区域有轻微的增加,而MGI平台则主要集中在高GC区域,在稍低质量样本中,这一趋势被进一步放大(图7)。小编愚见,在捕获实验中,是否增加预文库投入量,降低Post-PCR循环数以进一步提升相关表现。
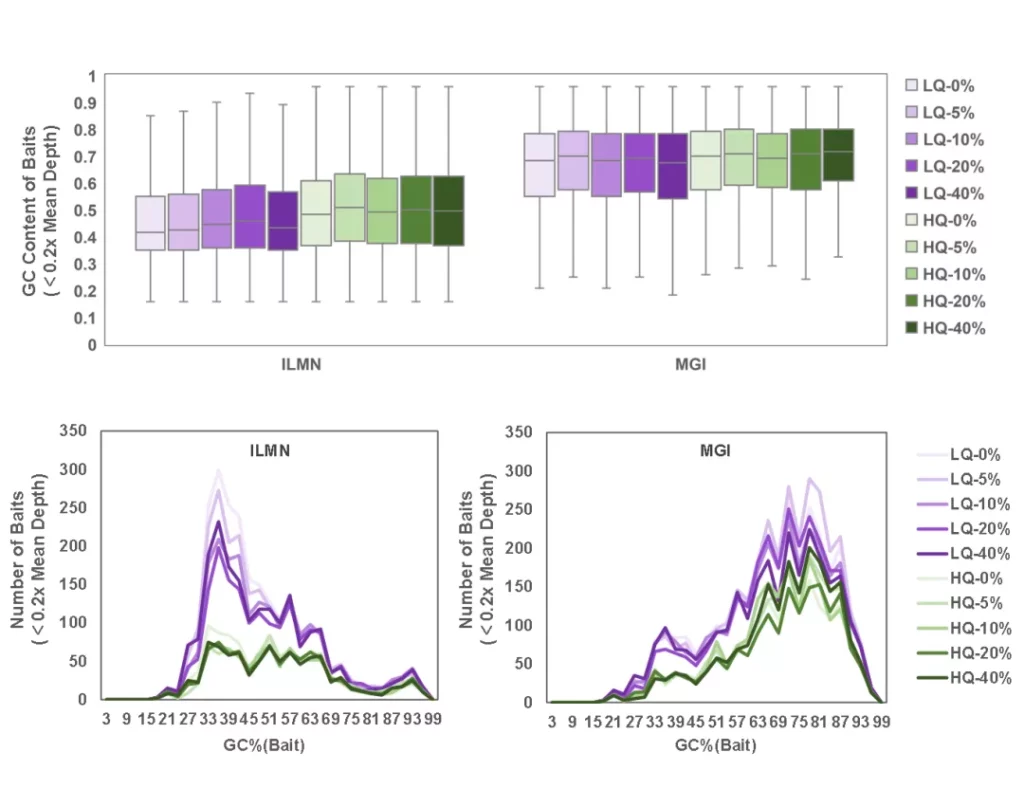
图7. TargetCap®Core Exome Panel v3.0低覆盖区域的平台分布差异
综上,我们欣喜的看到国产WES芯片已经可以完美适配Illumina Novaseq与MGI-2000平台,且性能表现优异。在下篇文章中,我们将给大家带来TargetCap Core Exome Panel v3.0在不同测序平台变异检测方面的内容,敬请期待。
参考文献:
[1] Reinert T , Henriksen T V , Christensen E , et al. Analysis of Plasma Cell-Free DNA by Ultradeep Sequencing in Patients With Stages I to III Colorectal Cancer[J]. JAMA Oncology, 2019, 5(8).[2] Reinert T , Henriksen T V , Christensen E , et al. Analysis of Plasma Cell-Free DNA by Ultradeep Sequencing in Patients With Stages I to III Colorectal Cancer[J]. JAMA Oncology, 2019, 5(8).[3] Parsons H A , Rhoades J , Reed S C , et al. Sensitive Detection of Minimal Residual Disease in Patients Treated for Early-Stage Breast Cancer[J]. Clinical Cancer Research, 2020, 26(11):clincanres.3005.2019.