NAR | TAPS+长读长测序技术破译DNA甲基化模式
- boke
- 2022-07-25
- 7:21 上午
在哺乳动物基因组中,DNA胞嘧啶修饰是调控基因时空表达的关键表观遗传机制。DNA甲基化有足够的潜力作为癌症的生物标志物,用于筛查、预后以及疗效监测。传统的短读长亚硫酸氢盐测序(BS-seq)被认为是甲基化分析的金标准。尽管BS-seq被广泛采用,但它有两个主要缺点:1、由于DNA脱嘧啶导致DNA严重降解,使得很难获得足够长的DNA片段进行长读长测序。2、由于未修饰胞嘧啶C-T转换,亚硫酸氢盐文库降低了序列复杂性。复杂性降低导致测序质量差、比对率低和基因组覆盖不均。
TAPS(TET-assisted pyridine borane sequencing)是通过TET1氧化酶将5mC和5hmC氧化为5caC,然后被吡啶硼烷还原为二氢尿嘧啶(DHU)。随后被扩增生成了C转化为T的PCR产物。与BS-seq相比,TAPS是非破坏性的,具有更好的测序质量、比对率和覆盖率。基于酶学方法的 DNA 甲基化测序技术 (EM-seq),使用TET将5mC转化为5caC,APOBEC3A催化胞嘧啶脱氨成尿嘧啶。与TAPS相比,通过将未修饰的胞嘧啶转化为胸腺嘧啶间接检测获得,这在得到的测序文库中导致了同样的低复杂度问题。
长读长测序技术为精确组装基因组、单倍型分型、串联重复序列和大片段结构变异解析提供了机会。然而PacBio SMRT测序对5mC的检测需要超高的序列覆盖率,Nanopore测序则需要已知甲基化状态的对照样本和复杂的计算分析。此外,SMRT和Nanopore甲基化测序都需要微克水平的非扩增DNA作为输入。鉴于TAPS和EM-seq方法都能在更温和的酶和化学条件下更好地保存较长的DNA片段,结合长读长测序和无亚硫酸氢盐方法将进一步提供高精度的长读长表观遗传测序。

为了进一步拓展长读长表观遗传测序的应用,2022年7月18日牛津大学路德维格癌症研究所宋春啸团队在《Nucleic Acids Research》上发表了题目为“Whole-genome long-read TAPS deciphers DNA methylation patterns at base resolution using PacBio SMRT sequencing technology”的研究,将TAPS方法与PacBio SMART测序相结合的全基因组长读长测序TAPS(wglrTAPS)方法,在全基因组尺度上精确检测长距离DNA甲基化。最新的PacBio SMRT测序准确度>99.9%。
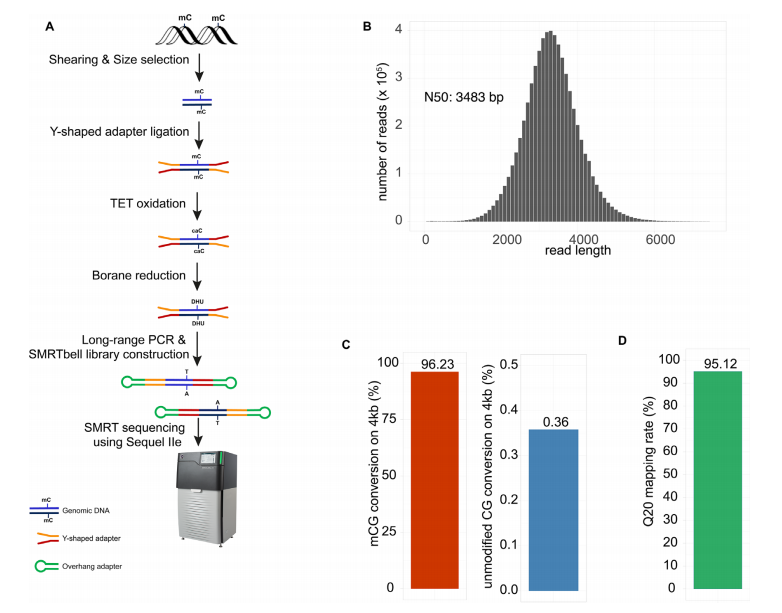
图1 wglrTAPS方法将TAPS与PacBio SMART测序相结合
为了评估wglrTAPS的性能,研究人员将wglrTAPS方法应用于小鼠胚胎干细胞(mESC),共生成了650万个N50为3.5kb的reads,并且当wglrTAPS测序深度为8.2×时,短读长TAPS与wglrTAPS在个体CpG水平上的相关系数仍达到0.68。并且发现很大比例的CpG位点,这是在之前深度为27.5×短读长TAPS中没有覆盖的。研究结果表明,wglrTAPS能够实现具有重复序列或sv的复杂区域的甲基化分析,而短读长TAPS无法实现。通过分析长读长序列数据,wglrTAPS能够剖析印记基因中等位基因特异性甲基化。此外,wglrTAPS还可以告知重复区域内的甲基化状态,这些都是短读测序方法难以解决的。
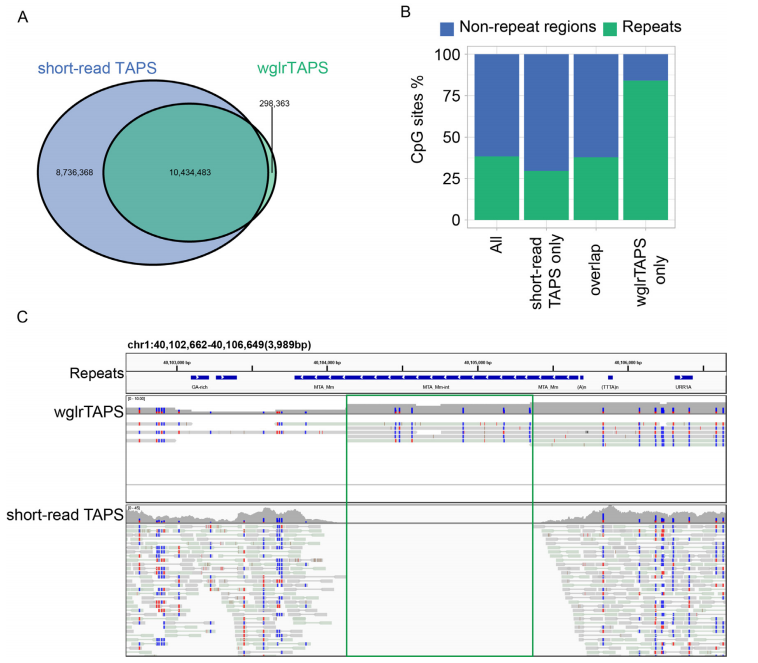
图2 短读长TAPS与wglrTAPS的CpG覆盖比较
该研究结合PacBio长读长测序技术开发了一种全新的全基因组长读长甲基化分析方法-wglrTAPS。该方法促进了三代测序技术在DNA表观遗传学中的应用,将TAPS的低损伤以及最低幅度改变基因组组成的优势与长读长相结合,wglrTAPS提供了整个基因组的长片段甲基化分析。同时,WglrTAPS可以广泛应用于其他临床材料,如已知长序列重复和印记基因的DNA甲基化异常与癌症等人类疾病。