技术小贴士 | PCR是偏好之源?对Capture而言并非如此
- boke
- 2022-06-23
- 7:21 上午
上一篇文章我们介绍了基于液相基因芯片的基因靶向测序技术中,探针富集的基本工作原理(技术小贴士|探针高温变性时间对捕获的影响)。预文库构建时,通常都会经过PCR扩增,每个Unique的DNA模板都存在大量拷贝;靶向捕获过程是用过量的探针对Unique片段扩增后的大量拷贝进行富集,确保“万无一失”(图1)。
如果我们采用PCR-free的预文库进行捕获,其实是去掉每个Unique模板的副本“保险”,即Unique模板没有大量拷贝存在,探针的亲和性偏好将被放大,其模板利用率和覆盖均一性将会受到影响。那么,PCR–free预文库的靶向捕获数据如何,小编就带大家一探究竟。
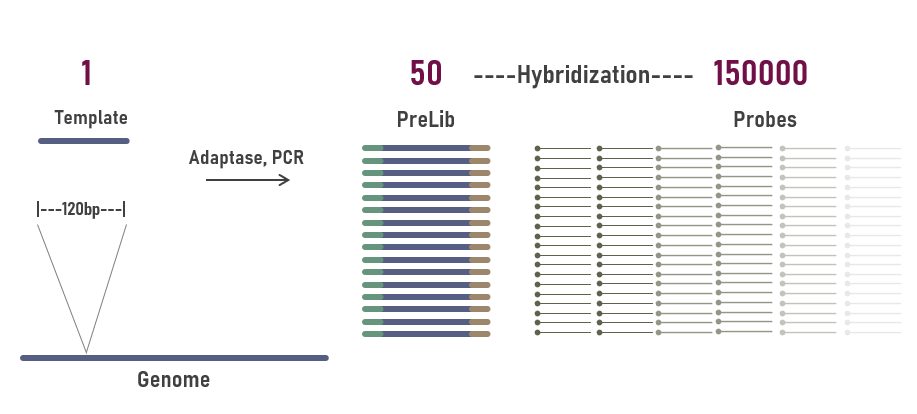
图1. 杂交捕获中,原始模板(Template)、扩增后预文库(PreLib)与探针(Probe)的分子数量示意图
使用50ng DNA投入量进行文库构建,连接Truseq接头(“大Y”),分别PCR 0、2、4和6个循环;为了更好的测试模板利用率表现,在捕获环节使用小尺寸Panel(<50Kb)进行捕获,这样能够较快达到文库的饱和深度,同时,将不同条件的预文库全部用于捕获,并在捕获后采用相同的PCR循环数以尽可能减小实验变量。此外,为了测试提高投入量能否改善PCR-free PreLib的相关表现,我们增加了200ng DNA Input的PCR-free PreLib的条件测试(图2)。

图2. PCR-free预文库捕获测试实验设计
如图3所示,在25000x理论深度条件下进行分析,50ng PCR–free PreLib捕获数据的Duplication已经达到近80%,明显高于其他建库条件;随着PCR循环数的增加,其Duplication逐渐降低,并在4-6个循环时不再降低,证明50ng PCR–free PreLib的捕获文库丰富度低于其他条件。我们也注意到,200ng PCR-free PreLib的Duplication表现与50ng PCR 4-6个循环的条件相近,说明提高DNA建库投入量,对于在PCR-free PreLib的捕获文库丰富度提高是有帮助的。
同样,在25000x理论深度条件分析的Unique Depth表现与Duplication较为一致,50ng PCR-free PreLib捕获的目标区域深度只有PCR 4-6个循环的一半左右,也就是说,采用PCR–free PreLib进行捕获将丢失大量的原始模板。
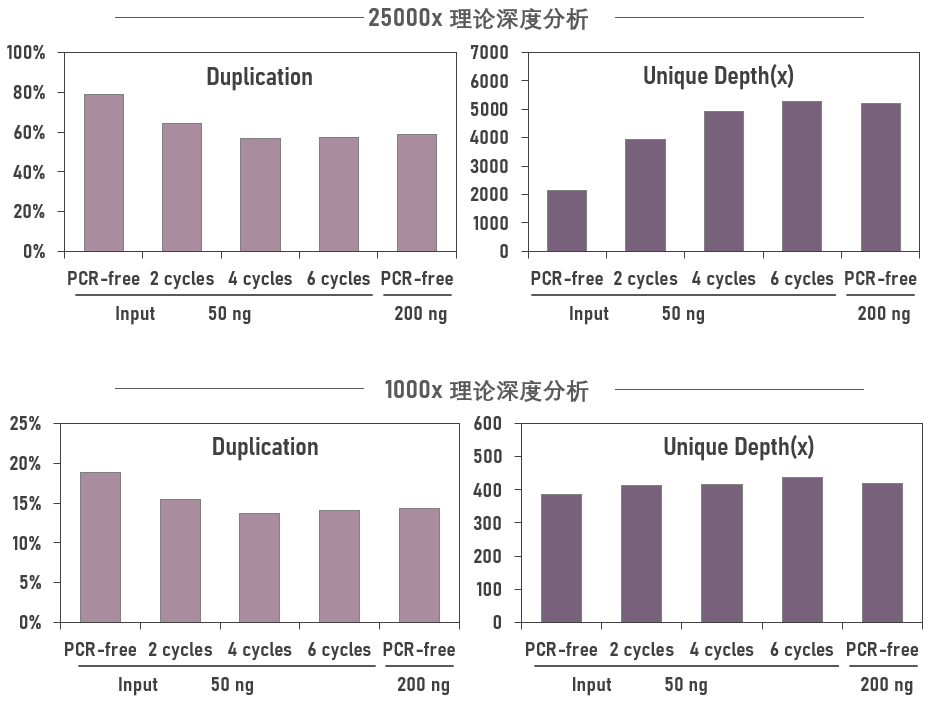
图3. 不同数据量条件下,PCR-free和PCR预文库的捕获数据(Duplication,Unique Depth)
此外,如果采用较低的测序深度进行分析(图3. 1000x理论深度分析),虽然上述不同条件的捕获数据表现趋势较为相似,但并不能观测到PCR-free PreLib捕获严重丢失原始模板的现象。
在覆盖均一性方面,0.2x Mean、0.5x Mean和Fold80参数的表现与Duplication和Unique Depth的趋势一致。不同的是,提高投入量后,虽然200ng PCR–free预文库的文库丰富度明显改善(图3),但在均一性方面,200ng Input并未明显提高其捕获覆盖均一性,50ng与200ng Input的表现相近(图4),说明对于50ng Input的PCR-free预文库,覆盖较差的区域即使提高投入量也效率有限,至少提高4倍至200ng是这样。
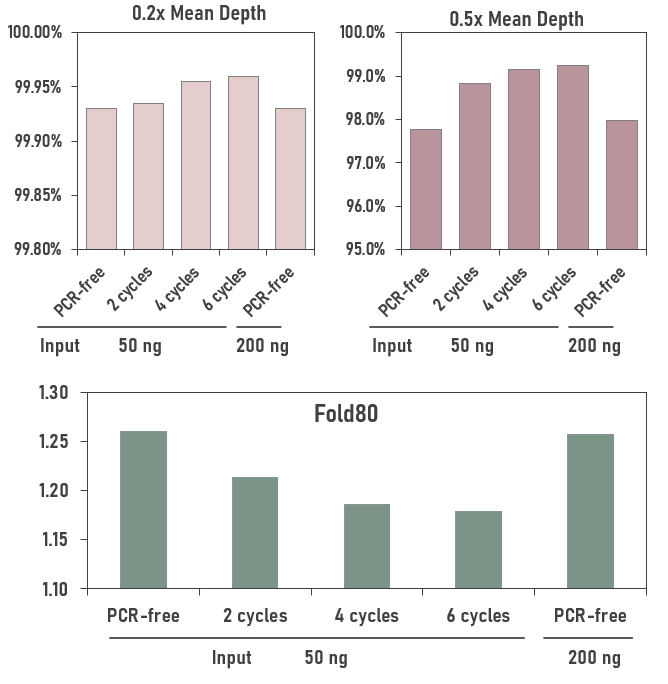
图4. 25000x理论深度条件分析PCR-free和PCR预文库的捕获均一性
综上所述,通过PCR增加原始模板的“拷贝副本”,可以有效提高捕获环节的模板利用率和覆盖均一性,因此,对于液相基因芯片捕获而言,常常产生偏好的PCR反而是“有益”的,特别是对于起始量较低,同时需要测序较高深度的样本,比如cfDNA。





