伯科甲基化Panel设计与应用
2021-03-10
DNA甲基化是在DNA甲基化转移酶(Dnmt)的作用下,甲基被选择性地添加到胞嘧啶的过程。该过程是一种重要的表观遗传学修饰,其不改变DNA序列,但能够调控基因的表达,参与早期胚胎发育、基因组印记、X 染色体失活、重复序列的沉默以及癌症的发生发展转移等多种生物学过程中,发挥了重大作用。通过甲基化测序有助于阐明这些重要生命过程背后隐藏的奥秘。
DNA甲基化作为一种重要的表观标志物,已被广泛应用于癌症早期筛查、良恶性肿瘤诊断、单基因遗传病诊断等研究领域。随着表观遗传学的发展,研究人员发现,获取基因组单片段、单碱基分辨率的甲基化数据,更有利于DNA甲基化标志物的发现。
目前,主要的高通量单片段(单倍型或者叫单体型,一个DNA片段上甲基化的组合)、单碱基分辨率的甲基化分析技术有三种(图1):
- 化学法-重亚硫酸盐测序(BS-Seq)
- 酶学法-EM-Seq
- 半酶学法-TAPS

图1. 三种高分辨率甲基化测序技术
BS-Seq是单片段、单碱基分辨率甲基化分析的“金标准”,作为化学法转化其转化效率高,但DNA损伤和偏好性是其应用的主要瓶颈。与之相比,酶学法和半酶学法甲基化测序技术对DNA造成的损伤小,其中,TAPS对基因组GC含量影响更小(技术介绍见文末)。因此,后两种技术越来越多的受到研究人员的青睐。
甲基化标志物的一大特点是甲基化位点数目较大,往往达到几千或上万个,靶向甲基化测序技术一次检测的甲基化位点多,覆盖均匀,较其他技术更加适合单片段、单碱基分辨率甲基化标志物的检测。
依托高通量合成平台,伯科生物自主开发的靶向甲基化测序技术适用于上述三种甲基化检测技术,可分别针对BS-seq/EM-Seq和TAPS转化设计相应的甲基化基因Panel。伯科甲基化Panel针对正负链均设计探针,能有效区分表观修饰和碱基变异,适用于多组学共检测。此外,数据表现优异,捕获效率高,覆盖均一性好。
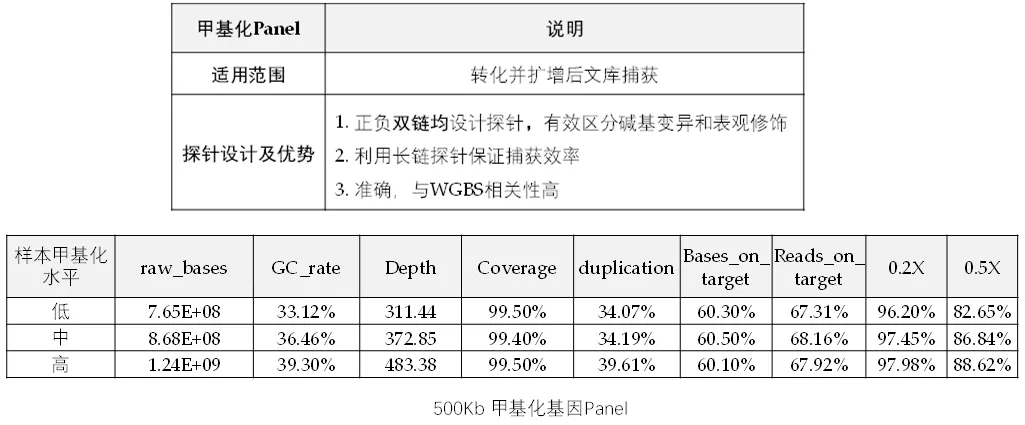
图2. 伯科甲基化捕获芯片数据表现
伯科的甲基化芯片有如下优势:
■ 适用上述三种方法处理并扩增后的文库捕获。
■ 针对正负链设计探针,获得丰富的双链信息。
■ 优异的捕获特异性、CpG 覆盖率与均匀性。
■ 偏好低,与WGBS相关性好。
■ 百款Panel设计经验,精心助力Panel设计。
■ HPLC高纯度质控,极速生产交付。
化学法-重亚硫酸盐测序
BS-Seq是单片段、单碱基分辨率甲基化分析的“金标准”,作为化学法转化其转化效率优秀,在99.5%左右,但高损伤是其应用的主要瓶颈[1]。为了保证后续文库的多样性,对DNA的起始量有较高的要求,所以在处理cfDNA样本这种起始量较低的样本时,BS的处理方法相对比较困难;再比如BS处理后片段断裂有序列特异性,以及对胞嘧啶的转化也并非是100%,这些缺陷也会造成后续甲基化分析时的偏差。

图3. BS前处理和后处理的文库制备方案的数据比较[2]
有研究报道了BS过程中不同的加热处理的温度、时间、变性方法等条件对后续测序结果的覆盖度、偏好性的影响,高温加热和高浓度的亚硫酸氢盐都会对DNA造成更严重的损伤[2]。所以BS和二代测序的结合,需要搭配合适的实验流程才能有较为理想的数据结果,对于Target-BS测序,伯科推荐采用Post-BS方法构建预建库,以提高模板丰富度。
酶学法-EM-Seq
NEB公司(New England Biolabs)开发了一种使用酶学转化替代化学处理,能同时检测5mC和5hmC的新方法——NEBNext酶学转化法甲基化建库试剂盒(EM-seq™)。
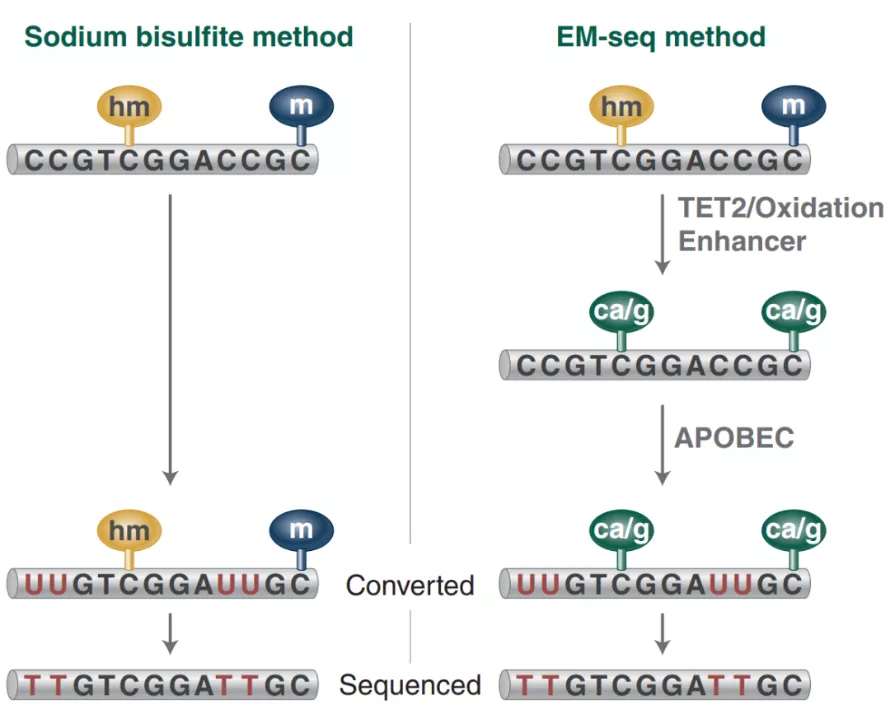
图4:亚硫酸氢钠转化与EM-seq原理比较(来源/NEB)
EM-seq中对DNA的酶处理分为两步:
第一步使用TET2酶以及氧化增强剂将5mC和5hmC氧化成5caC,保护经过修饰的胞嘧啶;
第二步利用脱氨酶APOBEC对胞嘧啶进行脱氨处理形成尿嘧啶,此步骤不会对5caC造成影响。
EM-seq流程对DNA更为温和,更大程度地减少了对DNA的损伤:
1、EM-seq转化后的DNA比重亚硫酸盐转化后的DNA更为完整,得到的文库中拥有更多更长的插入片段。
2、EM-seq流程中更温和的转化方式可保证获得高质量的DNA文库,继而进行高效扩增。因此,相较于WGBS,EM-seq可用更少的PCR循环实现更高的文库产量。
3、EM-seq文库显示了更均一的 GC 覆盖度,能比重亚硫酸盐文库检测到更多的CpG位点。并且在低测序深度下,EM-seq也能检测到更多的CpG位点。
4、EM-seq文库在不同起始量之间的相关性都很高,表明甲基化检测灵敏度不会随着起始量的减少而降低。
此外,EM-seq的转化结果与亚硫酸氢盐测序一致,故可采用同一套分析流程工具。
半酶学法-TAPS
牛津大学路德维格癌症研究所宋春啸和Benjamin开发的一种新的DNA甲基化测序方法:TET辅助吡啶硼烷测序(TET-assisted pyridine borane sequencing,TAPS)。TET酶(ten-eleven translocation:10-11转位酶)能够把甲基化的C碱基转化成羧基化的C碱基,接着在吡啶硼烷作用下把羧基化的C转化成二氢尿嘧啶。而非甲基化位点则在此过程中不改变。
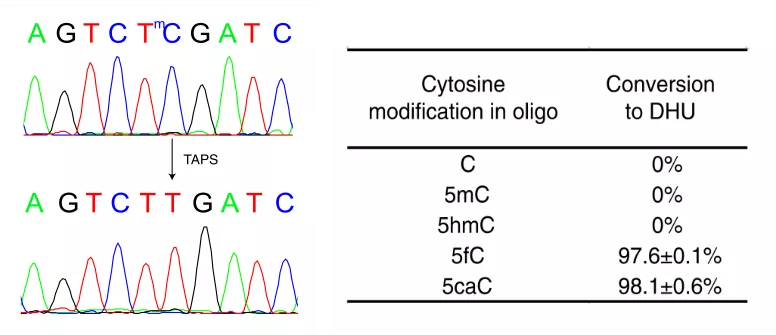
图5. Sanger测序结果显示,TAPS仅将5mC转换为T(左图);通过HPLC-MS / MS定量各种胞嘧啶衍生物的转化率(右图)。
通过这项技术,可以对比转化前后哪些C碱基被转化成了T,就能知道哪些C是被甲基化修饰。研究显示TAPS的假阴性率 2.7%-3.5%,假阳性率0.23%[3]。总结有以下优点:
1、与亚硫酸氢盐测序相比,TAPS数据的处理速度快了2倍
2、TAPS方法对DNA的破坏很小,可以保持10KB以上的长链DNA。
3、只要1ng的DNA量就能建库,相比重亚硫酸氢盐的方法(1ug DNA)更加具有样本普适性。
4、TAPS得到的DNA文库复杂度高(仅把占比很小的甲基化C进行了改变,对DNA的天然序列改变很小,碱基平衡),覆盖范围更均匀,定位效率更高,可以生成更精确的测序数据。此外,TAPS测序成本仅为WGBS的一半。
目前,TAPS可与多种下游分析技术兼容,包括但不限于甲基化敏感性PCR、限制性酶切、质谱、全基因组测序等,还可与其他测序方法结合,进一步降低成本,提高分辨率。
参考文献
[1] Tanaka, K. & Okamoto, A. Degradation of DNA by bisulfite treatment. Bioorg. Med. Chem. Lett. 17, 1912–1915 (2007).
[2] Olova N et al. Comparison of whole-genome bisulfite sequencing library preparation strategies identifies sources of biases affecting DNA methylation data[J]. Genome Biology, 2018, 19(1):33.
[3] Liu Y et al. Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution[J]. Nature biotechnology, 2019, 37(4): 424-429.