Genome Medicine| UMI+Amplicon+Nanopore技术路线,尝试解析LPA KIV-2内部变异与单倍型
- boke
- 2024-11-08
- 5:54 下午
脂蛋白(a)[Lp(a)]是导致动脉粥样硬化心脑血管疾病(ASCVD)的重要致病因素,我们每个人Lp(a)的浓度高低90%以上由遗传决定,这就意味着,心脑血管疾病易感性在很大程度上与Lp(a)的分子遗传特征相关。考虑到ASCVD在全球的高发病率,准确解析Lp(a)的遗传特征可以认为是人类基因研究中最重要的工作之一。
Lp(a)由类LDL颗粒与载脂蛋白(a)[Apo(a)]组成,其中,Apo(a)是Lp(a)浓度的决定性因素,由LPA基因编码。LPA基因的复杂性主要来自其VNTR(Variable number tandem repeats)区域的KIV-2结构域,高度同源的VNTR基本单元长度达到5.5Kb左右,拷贝数量可以达到40个,长度可超过200Kb,除了比对困难外,这个长度已经超过了现有检测技术的极限;此外,超长的VNTR还限制了全长LPA基因单倍型的解析,导致很难全面了解LPA的分子特征。
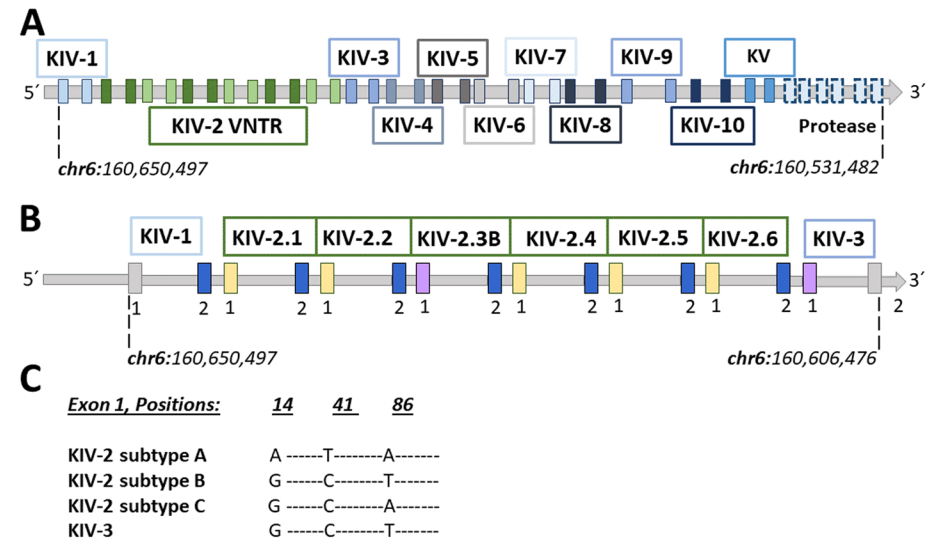
LPA基因结构示意图[1]
近期,Genome Medicine发表的一项研究[2]又将LPA基因的分子解析工作向前推进了一步,利用带有分子标签的扩增子技术搭配Nanopore长读长测序平台构建的管线,可以详细分析LPA基因KIV-2 VNTR中的嵌合序列变异、STR变异、不同KIV-2亚型、内部单倍型以及VNTR拷贝数量。
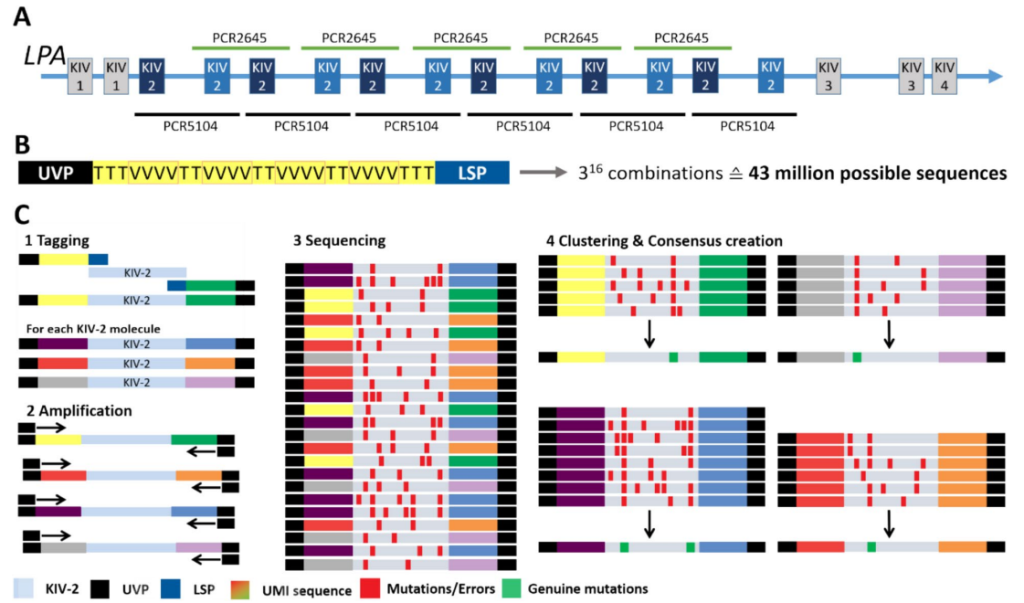
方法
基于扩增子的纳米孔测序与独特的分子标识符 (UMI-ONT-Seq) 在 KIV-2 VNTR 中检测 SNP、构建单倍型图、生成 VNTR 单元共识序列,以及通过覆盖率校正的单倍型定量来估算拷贝数的性能。使用15个人的样本和0.5%到5%的KIV-2质粒混合物作为验证集;然后,应用 UMI-ONT-Seq分析了48 个来自 1000 个基因组项目的多种族样本,提取了KIV-2 VNTR的单倍型,并对KIV-2VNTR中的短串联重复序列 (STR) 进行了大规模分析。
结果
UMI-ONT-Seq 检测到 KIV-2 SNP,变异水平低至 1%,具有高灵敏度、特异性和精确度 (0.977;1.000;0.993),并准确地获得了每个 VNTR 单元的完整单倍型。变异检测与NGS测序高度相关 (R2 = 0.983),在整个变异水平范围内没有偏好性。每个 KIV-2 单元的 6 个读长产生了 Q40 质量的序列。通过覆盖率校正的唯一单倍型计数确定的 KIV-2 重复数与ddPCR结果基本一致,其中 70% 的样本甚至落在 ddPCR 的狭窄置信区间内。随后,分析了 62,679 个 KIV-2 内的 STR 序列,并探索了五个不同祖源人群中的 KIV-2 SNP 单倍型模式。
结论
UMI-ONT-Seq 能够准确获取 SNP 单倍型,并精确量化复杂 KIV-2 VNTR 区域每个重复单元的 VNTR 拷贝数,涵盖多个祖源人群。这项研究利用 KIV-2 VNTR,提供了一种新颖而有效的工具,用于大规模全面表征这类携带VNTR区域的医学相关的复杂基因组区域。
参考资料
1. Di Maio S, Zöscher P, Weissensteiner H, et al. Resolving intra-repeat variation in medically relevant VNTRs from short-read sequencing data using the cardiovascular risk gene LPA as a model. Genome Biol. 2024;25(1):167.
2. Amstler S, Streiter G, Pfurtscheller C, et al. Nanopore sequencing with unique molecular identifiers enables accurate mutation analysis and haplotyping in the complex lipoprotein(a) KIV-2 VNTR. Genome Med. 2024;16(1):117.
3. Nordestgaard BG, Langsted A. Lipoprotein(a) and cardiovascular disease. Lancet. 2024;404(10459):1255-1264
推荐阅读
这是示例文本,单击 “编辑” 按钮更改此文本。


