Nature Genetics | 机器学习定量描述冠心病表型,深度挖掘全外显子数据中冠心病罕见致病变异,促进冠心病致病机制研究
- boke
- 2024-10-25
- 6:01 下午

摘要
冠状动脉疾病(CAD)是由多种风险因素和病理过程组成的一类疾病,通过简单的是否来作为疾病判定,无法准确描述CAD的进展和严重程度,因此难以发现在人群中发生频率较低的罕见基因变异。
利用机器学习对电子健康记录中的临床数据构建CAD的连续表型评价系统(ISCAD),能够发现该疾病谱上的疾病进展、严重程度和漏诊情况,可能增强对CAD的遗传发现。
在本研究中,测试了英国生物库(UKB)、All of Us研究计划和BioMe生物库中罕见和超罕见编码变异(coding variants)与冠状动脉疾病表型评分-ISCAD的关联。
研究结果显示,有17个基因发现了关联;其中,14个基因显示出至少中等程度的先验遗传、生物和/或与CAD相关的临床支持。此外,还观察到超罕见编码变异在321个CAD强相关基因中出现了聚集,提示还有更多超罕见变异的关联有待发现。这些结果扩展了我们对CAD遗传病因的理解,并说明了多标志物如何增强对复杂疾病的遗传关联研究。
引言
冠状动脉疾病(CAD)是全球发病率和死亡率最高的疾病。CAD具有强烈的遗传因素,估计遗传率为40%–60%。对超过一百万参与者的大规模全基因组关联研究(GWAS)已识别出321个与动脉粥样硬化相关的基因。
大多数易感位点由位于非编码区域的常见(MAF >0.05)单核苷酸变异(SNVs)驱动,因此识别潜在的致病基因很困难。相反,罕见(MAF ≤0.01)和超罕见(MAF ≤0.0001)错义或蛋白质截断变异(PTVs)对蛋白质功能有直接影响,这可以揭示基因在疾病病理生理学中的作用并揭示潜在的治疗靶点。
利用NGS全外显子测序数据(WES)进行的罕见变异关联研究能够调查人类基因组中所有编码蛋白质基因的罕见和超罕见编码变异。迄今为止,针对CAD的大规模外显子测序研究很少,并且在发现CAD的罕见编码变异方面成功有限。
例如,一项针对心肌梗死(MI)的WES测序研究,包括了4703例病例和5090例对照,发现了低密度脂蛋白受体(LDLR)和载脂蛋白A-V(APOA5)基因中存在罕见编码变异。另一项针对CAD的WES测序研究包含7671名汉族个体,识别出LDLR和QTRT1基因中的罕见编码变异。最近的一项基因测序研究发现NOS3基因中的罕见编码变异。
CAD的亚最佳表型可能限制了在与电子健康记录(EHR)相关的生物库中发现罕见变异。定义疾病案例和对照的标准方法依赖于诊断编码。这可能导致简单的判断是或否患有CAD,无法正确分类不同疾病严重程度或亚临床动脉粥样硬化的个体。过于简单化的CAD表示可能引入噪音,最终限制遗传关联测试的统计效能。由于CAD在普通人群中的低患病率,统计效能不足的问题进一步加剧。因此,需要新的表型方法来增强在基于EHR的遗传研究中进行的罕见变异关联研究的基因发现努力。
CAD自然存在于一个疾病谱上,因为狭窄和斑块组成的变化导致MI和死亡风险的差异。这个疾病谱由一系列风险因素和病理机制组成,这些都可以通过EHR中的临床数据捕捉。因此,CAD的连续表示可以在对照组内区分风险,考虑疾病进展和/或严重程度的不同程度,并识别误分类的个体。为此,本研究引入了一种基于机器学习的冠状动脉疾病表型评分(ISCAD),该评分是利用EHR中的临床数据构建的。ISCAD是根据一年CAD风险预测模型进行调整的。重要的是,该研究展示了ISCAD与疾病进展、严重程度和死亡率的相关性,并揭示了漏诊情况。
在该研究中,假设罕见和超罕见编码变异与ISCAD相关,这是对CAD谱的整体表示。为了评估这一假设,研究人员测试了在UKB、All of Us研究计划和BioMe生物库中604,915名个体的WES数据中罕见和超罕见编码变异与ISCAD的关联。通过评估这些变异与67个生物标志物特征、461个表型编码和动脉粥样硬化的临床表现的关联,调查了17个在外显子组范围内的显著变异和基因的作用。此外,还评估了这些变异和基因与CAD的GWAS中识别的基因组位点的重叠。
研究发现,已知和新发现的动脉粥样硬化生物过程相关的基因集中聚集了超罕见编码变异。最后,研究还展示了在由321个CAD相关基因组成的基因集中超罕见编码变异的富集,表明还有更多具有罕见编码变异的基因待发现。
结果
研究人群描述
使用来自UKB的502,505个个体的EHR数据、来自All of Us研究计划的113,575个个体以及来自BioMe生物库的43,744个个体来构建每个生物库中ISCAD的机器学习模型。
使用来自英国生物库的464,416个个体、All of Us研究计划的106,926个个体、BioMe生物库样本1中的22,515个个体,以及BioMe生物库样本2中的额外11,058个个体的外显子序列数据进行关联测试。
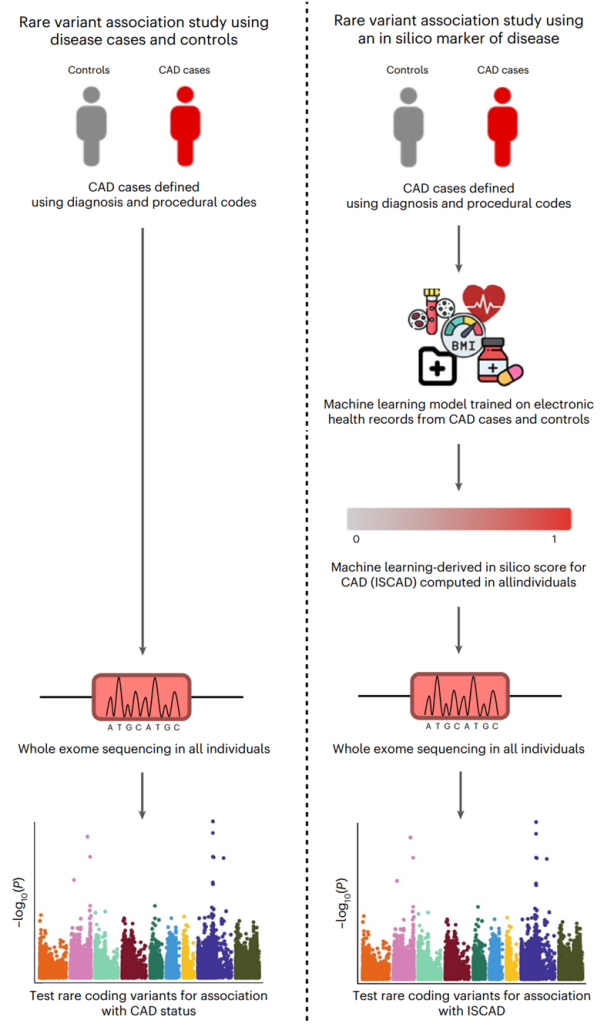
图1.冠心病机器学习表型评价模型-ISCAD与传统CAD表型在研究罕见变异的区别的示意图
ISCAD的构建
根据先前描述的工作流程进行了调整,以考虑所有个体和临床特征;确保模型在不同EHR系统和研究样本之间具有普遍适用性。
使用来自49,254、17,049和6,694例新发和现患CAD病例的EHR数据,分别在英国生物库、All of Us研究计划和BioMe生物库中拟合机器学习模型。这三种模型在CAD病例与对照之间显示出良好的区分能力,AUROC(receiver operating characteristic)分别为0.81、0.86和0.89。

图2. 三个数据库的ISCAD准确性[UK Biobank(A)、All of Us(B)和BioMe(C)]
模型考虑的已知CAD临床特征包括年龄、糖化血红蛋白A1c、低密度脂蛋白胆固醇(LDL-C)、高血压诊断和抗凝药物处方。在所有三个生物库中,CAD病例中的ISCAD分布与MI病例的分布高度重叠,而对照组的ISCAD则低于CAD病例。此外,ISCAD在所有生物库中与CAD进展、严重程度和漏诊相关,这些结果重复出了之前研究的结果,表明ISCAD有效捕捉了队列的潜在CAD状态,作为遗传关联分析的定量表型。
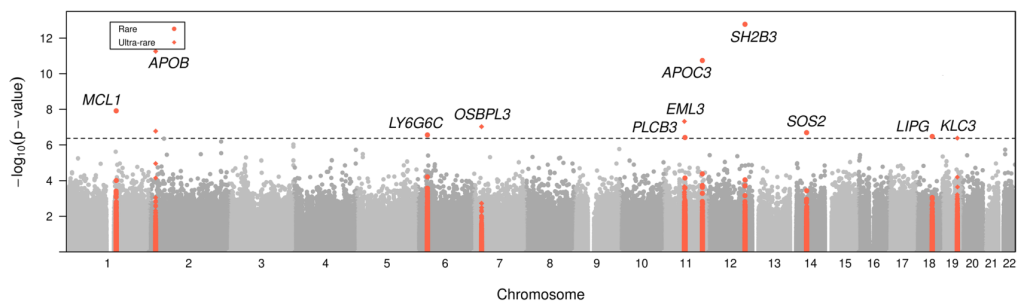
图4. 罕见变异关联的曼哈顿图
其中六个变异会增加CAD的风险,分别为rs72650673(SH2B3, E400K)、rs5742904(APOB, R3527Q)、rs142066904(APOB, R439*)、rs11575848(LY6G6C, R103Q)、rs145502455(PLCB3, V806I)和rs1353787944(KLC3, Q86K)。
另外六个变异则提供了对CAD的保护,分别为rs138326449(APOC3, IVS2+1G>A,)、rs11580946(MCL1, A227V)、rs1802916301(EML3, G595S)、rs367606248(OSBPL3, R506G)、rs72681869(SOS2, P191R)和rs77960347(LIPG, N396S)。
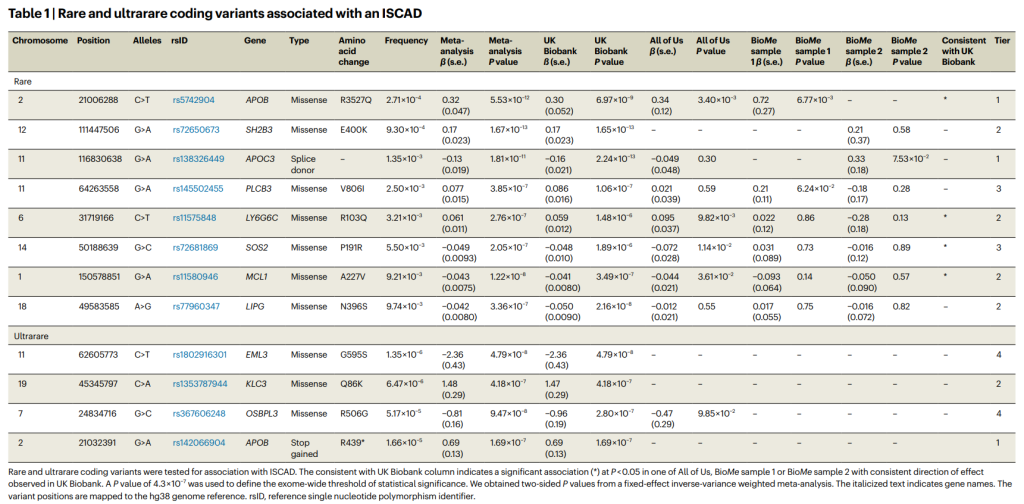
表1. 罕见与超罕见编码变异与ISCAD的关联
超罕见编码变异的关联发现受到统计功效不足的限制,因为携带次要等位基因的个体数量较少。因此,次要等位基因计数(MAC)非常低的单个变异无法单独进行测试。相反,可以通过聚合MAC <5的超罕见PTVs和有害的错义变异进行基因级别的测试。
对17,883个基因中MAC <5的超罕见编码变异进行了ISCAD的关联测试。识别出七个基因,分别为HECTD4、LDLR、GCK、PPARG、ABCA1、ACVR1和APOB(表2)。其中,只有APOB在单个变异分析中被识别。总的来说,从变异和基因级关联测试中识别出总共17个与ISCAD相关的基因。
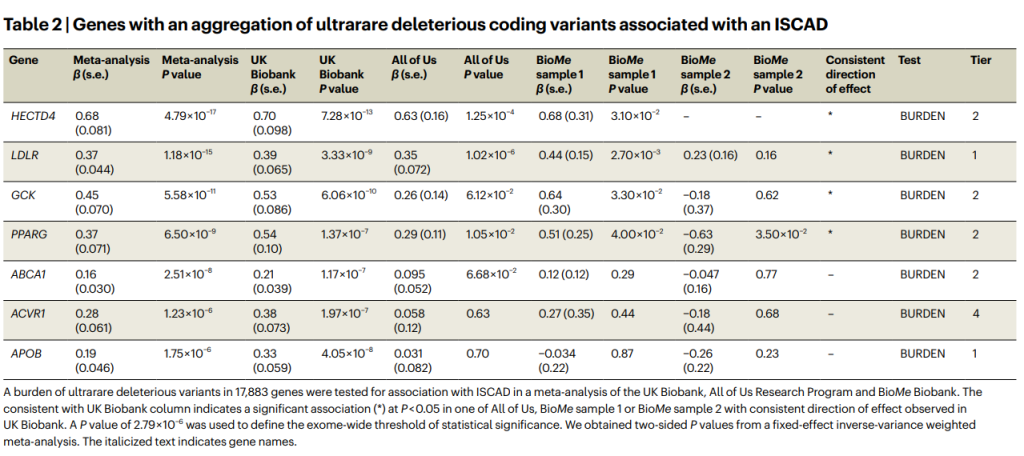
表2. 聚集超罕见有害编码变异的基因与ISCAD的关联
已知的CAD罕见编码变异关联
研究还评估ISCAD是否能够重现已知的CAD罕见编码变异关联。在UK Biobank的464,416个个体的WES数据中测试了ISCAD与7个基因(ANGPTL4、APOB、APOC3、LDLR、LPL、NPC1L1和PCSK9)的变异和基因水平关联。与常规二元CAD表型相比,ISCAD在六个(85.71%)变异水平和七个(100%)基因水平测试中显示出等同或更强的关联性。
值得注意的是,ANGPTL4、APOB、APOC3和LPL的关联在变异和基因水平分析中均达到了外显子范围的显著性,而常规二元CAD表型则未达到。此外,大多数与ISCAD的罕见变异关联在方向上与常规二元CAD表型的遗传关联发现一致。
我们观察到一个显著例外,即超罕见的APOB有害变异(仅由PTVs组成)在ISCAD中增加风险(APOB,β=0.33,P=4.05×10−8)。此外,错义变异rs11591147(PCSK9 R46L,β=−0.00086,P=0.91)在ISCAD中没有显示关联,尽管它在CAD状态上已知具有保护作用。
值得注意的是,研究揭示了APOB和PCSK9这两个基因的生物学复杂性,对CAD可能具有相互抵消的效应:APOB PTV变异对家族性低β脂蛋白血症和肝脂肪变性有影响,但降低LDL-C效应,而PCSK9则与糖尿病相关,但也降低LDL-C效应。
ISCAD基因的先前证据
为了评估ISCAD基因在CAD中的作用,我们定义了一个分层证据分类框架,根据与CAD相关的先验遗传、生物和临床证据对基因进行优先排序。考虑了9条独立证据,将这17个ISCAD基因划分为4个层级(图5)。
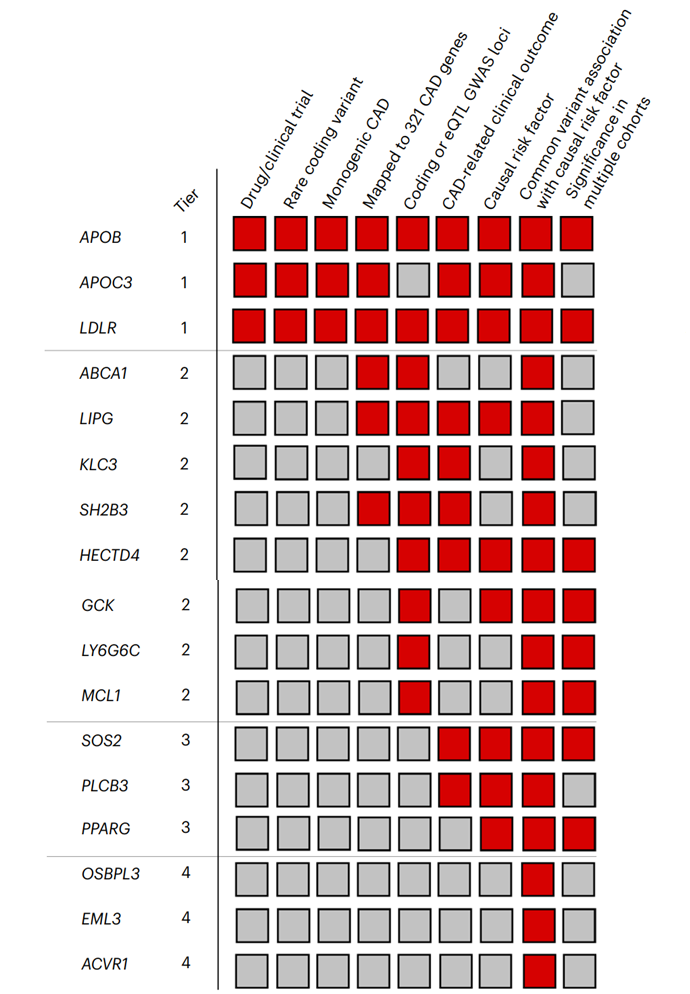
第一层级将具有强有力证据支持其在CAD中作用的基因归为一类。这些基因有临床试验显示其在CAD上是有效的药物靶点,或者与CAD有已知的罕见编码变异关联和/或单基因CAD。共有3个(17.65%;APOB、APOC3和LDLR)ISCAD基因被归入第一层级。
第二层级将基于以往GWAS对CAD的中等强度遗传证据的基因归为一类,这些基因要么在最近的综述中比对到321个CAD基因,要么在目前最大的CAD GWAS中有编码变异和/或表达数量性状基因组信号(P<10−6)。总体而言,8个(47.06%)ISCAD基因被归入这一层级。
第三层级则将支持其在CAD中作用的中等遗传证据基因归为一类,包括与因果CAD风险因素(即LDL-C、甘油三酯、脂蛋白(a)、体重指数、2型糖尿病和高血压)相关的罕见编码变异关联或与CAD相关的临床结果(即MI、心律失常、心力衰竭、动脉硬化指数和左心室射血分数)关联。总体而言,3个(17.65%)ISCAD基因被归入第三层级。
第四层级的基因展示了额外的CAD遗传证据。这些基因通过与因果CAD风险因素的全基因组显著关联(P<5×10−8)或在我们的分析中多个队列的显著性得到支持。被归为第四层级的基因可能暗示CAD风险的替代途径,例如OSBPL3与红细胞计数、EML3与血清Clara细胞16-kDa蛋白水平、ACVR1与网织红细胞计数的关联(补充表28)。有关每个基因的分子功能的描述,请参见补充说明。总之,17个ISCAD基因中有14个(82.35%)显示出中等到强的独立证据支持其在CAD中的作用。
ISCAD基因和其它指标的关系
接下来,研究分析了ISCAD基因与其他表型的关联。利用英国生物库中46万多人的电子病历数据,研究了19个基因(包含12个ISCAD变异体和7个ISCAD基因)与67个生物标志物和461个表型编码的关系。结果显示,10个ISCAD基因存在与58个生物标志物相关的罕见基因变异 (P<7.46×10⁻⁴),以及与五个表型编码相关的罕见基因变异 (P<1.08×10⁻⁴)。
其中,5个基因(APOC3、APOB、PLCB3、SH2B3、SOS2和LIPG)与脂类和脂蛋白代谢有关,4个基因(APOC3、SH2B3、SOS2和LIPG)与炎症标志物有关。有意思的是,LIPG基因的rs77960347变异不仅与高密度脂蛋白胆固醇、低密度脂蛋白胆固醇和甘油三酯升高有关,还与缺血性心脏病风险降低有关;而SOS2基因的rs72681869变异则具有预防高血压的作用。此外,另有6个ISCAD基因显示出超稀有基因变异的富集,这些变异体与32个独特的生物标志物和五个独特的表型编码相关。5个基因(ABCA1、APOB、GCK、LDLR和PPARG)中的超罕见有害变异体与脂类和脂蛋白代谢有关,但与炎症标志物无关。
已知CAD基因中超罕见突变的贡献
为了理解已知CAD基因中罕见编码变异的贡献,测试了ISCAD与321个CAD基因中超罕见有害变异的聚合关联。结果显示,所有321个CAD基因中聚合的超罕见编码变异存在过剩,而与超罕见同义变异没有关联。在去除本研究中发现的七个已知基因后,仍然观察到强关联。这些结果表明,其他CAD基因中的罕见和超罕见编码变异有待发现。
讨论
该项使用来自UK Biobank、All of Us研究计划和BioMe Biobank的四个独立研究样本的临床数据构建的基于机器学习的CAD连续表型与罕见变异关联进行荟萃分析,识别出17个基因中的罕见和超罕见编码变异。
该研究不仅发现了调节CAD风险因素的基因中的罕见和超罕见编码变异,如脂质代谢(ABCA1、APOB、APOC3、LDLR和LIPG)、2型糖尿病(HECTD4、GCK和PPARG)、高血压(PLCB3和SOS2)和炎症(SH2B3),还有涉及其他机制,如氧甾醇运输(OSBPL3)、有丝分裂纺锤体组装(EML3)、微管运输(KLC3)、抗凋亡(MCL1)和信号转导(ACVR1和LY6G6C)。使用分层框架考虑了多条独立证据来评估这些基因在CAD生物学中的作用,识别出14(82.35%)个ISCAD基因具有至少中等的先验遗传、生物和/或临床证据。
研究人员认为,使用疾病表型评分模型(如ISCAD)可能捕获额外的疾病病理生理学进展,这在理论上可以揭示在遗传关联研究中调节此类过程的额外遗传机制。
总之,该研究识别出与ISCAD相关的17个基因中的罕见编码变异。这些遗传关联增强了我们对CAD生物学的理解,超越了脂质代谢和标准风险因素。重要的是,这项研究提供了概念验证,表明基于机器学习的复杂疾病量化表型描述可以增强罕见编码变异的遗传发现。
上述ISCAD相关的17个基因以及4个已知CAD基因在伯科Core Exome Panel v3.0与晓芯甘基因芯片中均已涵盖,为冠心病相关科研与临床转化提供积极助力。
参考资料
1.Rämö J, Jurgens S J, Kany S, et al. Rare Genetic Variants in LDLR, APOB, and PCSK9 are Associated with Aortic Stenosis[J]. Circulation, 2024.
推荐阅读
这是测试文本,单击 “编辑” 按钮更改此文本。


