cfDNA片段长度研究进展
- boke
- 2023-01-17
- 7:07 上午
细胞游离DNA(Cell-free DNA,cfDNA)是一种片段化的DNA混合物,由多种身体组织释放的DNA分子组成;cfDNA可以用于监测多种生物学过程,并且已经在胎儿、肿瘤和移植器官产生的cfDNA分子中得到证实。cfDNA标志物为无创产前检测(NIPT)和癌症检测(液体活检)提供了重要的技术路径,NIPT在临床实践中成功地被全球采用,持续的研究工作已经扩展到无创癌症检测,并尝试将“NIPT模式”运用于肿瘤学研究中。
对cfDNA生物学特性的进一步了解往往会催生新的诊断工具。除了cfDNA片段本身序列的变异特征外,其片段化特征也非常有趣;cfDNA的片段化呈现出远超预期的多维度特征,事实上,cfDNA的片段化并非是随机的,而是包含了其来源组织的信息;此外,cfDNA的片段化特征还有可能作为不同组学之间的桥梁,跨组学的对基因表达(转录组学)和碱基修饰(表观组学)进行推演。目前,多种cfDNA片段化特征已经用于NIPT、癌症和器官移植评估的研究,如片段长度(fragment sizes)、片段末端(fragment ends)、末端基序(end motifs)等[1]。2022年12月,Jiang等对相关领域的最新进展进行了回顾总结,本文对cfDNA片段长度特征的研究进展进行介绍。
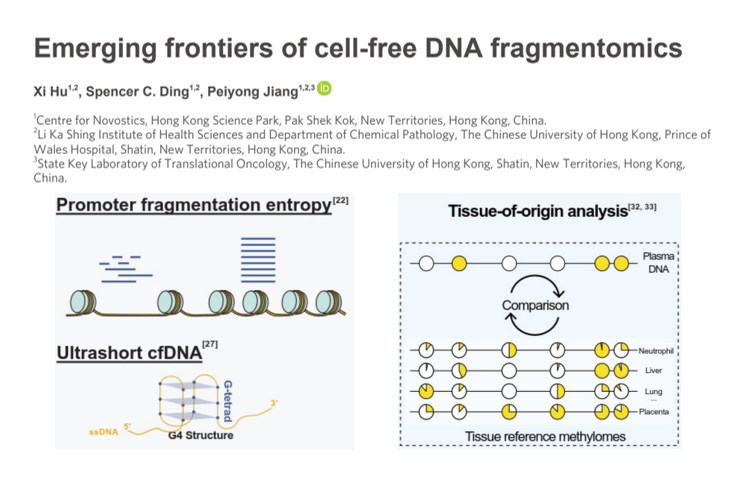
1998年,cfDNA分子的大小在凝胶电泳中被揭示,显示约180 bp倍数的梯状带。但凝胶电泳的分辨率相对较低,无法精确测量cfDNA分子的大小。研究人员试图通过设计不同大小的扩增子,使用qPCR技术来测定不同大小范围内cfDNA的相对数量,还有就是通过电子显微镜测量cfDNA分子大小。然而,无论是qPCR还是电子显微镜都无法对cfDNA的大小特征进行全基因组范围内的研究。
随着高通量测序技术(NGS等)的出现,对于源自几乎任何基因组位置的cfDNA分子,都可以在单碱基分辨率下推断出片段大小。NGS技术通常采用从两端对cfDNA分子进行测序(即配对末端测序),通过成对的末端测序Reads与参考基因组比对来确定片段的坐标及大小。NGS技术的测序总长度往往≤500bp并且只能获得碱基序列信息,因此,对于较大的cfDNA分子,中段序列信息是缺失的,同时,表观遗传信息也将丢失。长读长高通量测序技术的逐步成熟弥补了NGS的上述不足,Kb级别的长链cfDNA特征正在被揭示。

图1 cfDNA片段分析技术
2010年,Lo等人使用NGS技术破译了孕妇血浆DNA中胎儿和母体cfDNA分子的片段特征。在胎儿和母体cfDNA大小分布中,都在166 bp处有一个主峰,并在小片段区域伴随10bp的起伏波动,由于DNA分子与组蛋白循环缠绕形成核小体,暗示cfDNA分子片段化特征很可能与核小体结构有关。胎儿cfDNA分子的大小分布在143 bp处显示出第二个主峰,这一现象表明,胎儿cfDNA分子通常比母体cfDNA分子短。Lo等人推测,胎儿cfDNA分子的长度缩短可能部分归因于胎儿基因组受到组蛋白保护作用减弱,约20bp Linker会被优先切割[2]。因此,cfDNA片段是非随机的,并且与起源组织中的核小体结构相关。许多其他研究也再现了具有不同病理或生理状态的受试者血浆中cfDNA的大小分布特征,包括不同癌症、器官移植患者以及自身免疫性疾病患者。
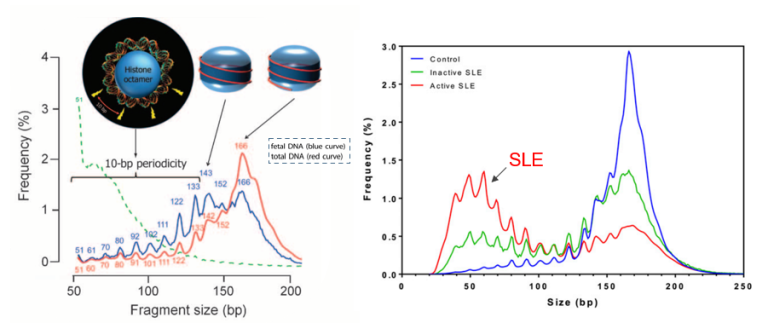
图2 cfDNA在生理和病理条件下长度分布
因此,不同组织来源的cfDNA存在长度差异的生物学现象被用于NIPT和肿瘤检测。2014年,Yu等人的研究表明,基于cfDNA的长度差异检测18和21三体的NIPT结果,与基于计数法结果完全一致,灵敏度与特异性均为100%[3]。同时,研究表明,癌症患者血浆中肿瘤产生cfDNA大小特性可以标志其来源,并有助于提升癌症检测性能。Jiang等人指出,利用肿瘤cfDNA更小的特性能够在肝癌患者中更高效地检测肿瘤产生的体细胞突变,PPV达到85%[4]。为了区分cfDNA的序列变异是否来自肿瘤,往往需要配对验证白细胞是否存在克隆造血变异,Marass等人验证了使用片段大小来区分克隆造血和肿瘤特异的体细胞变异的可行性[5]。Mouliere等人报道,通过利用cfDNA分子的数量,以及片段大小特征,癌症患者和健康人群的区分程度得到改善,AUC达到0.91(不采用片段大小<0.5)[6]。
2019年,Cristiano等人进一步开发了一种包括cfDNA长短变化参数在内的全基因组cfDNA片段化模式检测技术(DELFI),在分析236名乳腺癌、结直肠癌、肺癌、卵巢癌等多种癌症患者和245名健康个体后,研究结果显示该技术灵敏度范围为57%至99%,特异性为98%,其中75%的患者可以定位肿瘤[7]。2022年,DELFI技术性能进一步在肝癌临床队列中进行验证,该研究对自不同地区的肝癌(HCC)以及具有不同肝癌风险的非肝癌个体(724例)的血浆cfDNA进行了DELFI分析。研究结果显示,该技术检测癌症的灵敏度在平均风险人群中为88%,特异性为98%,在高危人群中为85%,特异性为80%,表明DELFI是一种检测HCC的可靠方法[8]。
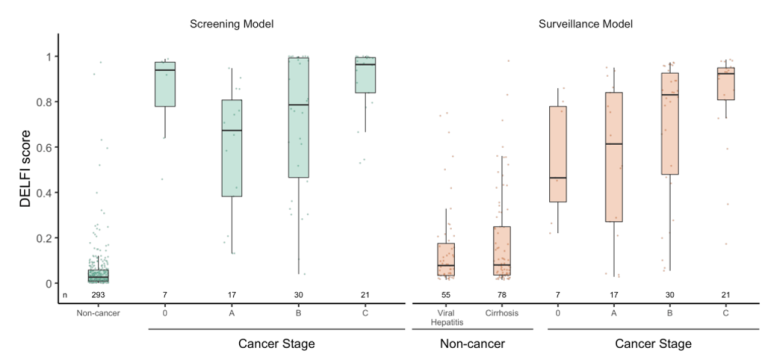
图3 利用DELFI评分检测肝癌
核小体在基因组DNA上的密度分布不同是导致cfDNA片段分布差异的重要原因,而通过对cfDNA进行高通量测序,分析基因组区域的深度覆盖分布,可以清晰的构建出核小体的分布模式;测序深度高意味着该区域受到了核小体保护,未被核酸酶切割,测序深度低则表明该区域可能无核小体占位,更容易被核酸酶降解。
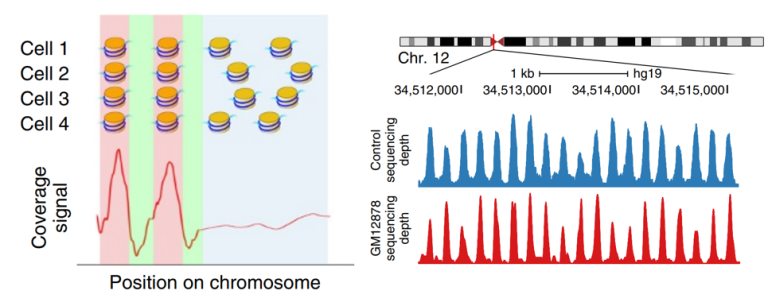
图4 核小体位置与测序覆盖(Coverage)的关系(左),右图显示了极端核小体占位对测序深度影响的示例
核小体的“足迹”深刻影响着基因调控,基于MNase方法,已经鉴定出大量在基因启动子区域的特定核小体分布模式。在基因的转录被激活时,转录起始位点(Transcription Start Sites ,TSS)的上游150bp存在一段核小体缺失的区域,被称为NDR(Nucleosome-Depleted Region),这段区域有助于庞大的转录复合物的进入,同时,NDR上下游的核小体被严格固定。相较而言,非激活的启动子的NDR既不表现出核小体缺失,也没有上下游核小体的严格占位[9、10]。
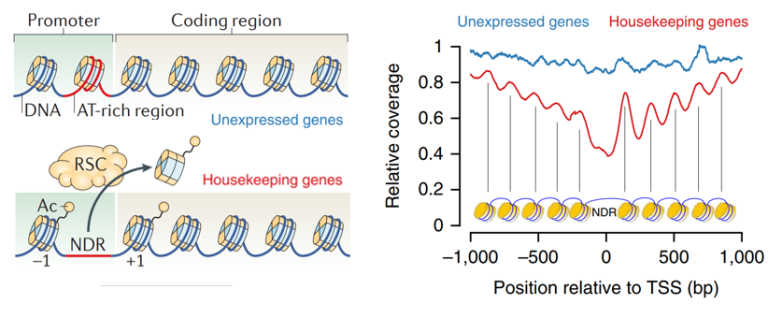
图5 基因的转录表达改变了核小体占位模式(左),并通过测序深度覆盖反映出来(右)
高转录活性区倾向于核小体占位降低,该区域DNA失去组蛋白保护,导致更多的随机断裂和更高的cfDNA片段长度变异性。2022年4月,Esfahani等人的研究证明,使用转录起始位点(TSS)周围的cfDNA片段的大小变化有助于预测基因的表达水平[11、12]。他们采用PFE(Promoter Fragmentation Entropy)量化源自启动子区域(TSS ±1Kb)的cfDNA片段的这种可变性,研究结果显示,PFE与RNA表达水平密切相关。为了进一步评估PFE的临床效用,Esfahani等人将具有肿瘤特异性表达基因的启动子作为生物标志物,在cfDNA中对这些标志物区域进行靶向深度测序,然后评估其PFE(EPIC-seq (epigenetic expression inference from cell-free DNA-sequencing))。该方法可以用于癌症检测、癌症分型和免疫治疗监测。基因启动子区域的片段大小变异的测量可以为癌症检测提供无创的生物标志物。需要注意的是,基于PFE的EPIC-seq数据分析发现的癌症患者90%的处于晚期(III或IV期)。

图6 通过靶向深度测序评估NDR区域的PFE,进而推断基因表达
此外,许多最近的研究表明,cfDNA片段化模式可以作为预测治疗结果的生物标志物,如鼻咽癌(NPC)的复发[13]、弥漫性大B细胞淋巴瘤(DLBCL)的治疗结果[14]和免疫疗法的应答[11]。鼻咽癌(NPC)患者在初次治疗结束时,通过qPCR定量检测血浆EB病毒(EBV)DNA是一个可靠的预后指标。然而,高达40%的患者在治疗后出现疾病复发,而血浆EBV DNA却检测不到。针对整个EBV基因组的靶向测序能够更全面和无偏好地检测血浆EBV DNA,并允许使用其他cfDNA参数,如片段大小作为生物标志物。因此,Chan等人验证了血浆EBV DNA靶向测序是否可以更准确地预测鼻咽癌患者的治疗结果。采用EBV DNA靶向测序分析769例放疗后6-8周的IIB-IVB期鼻咽癌患者的血浆样本,结果显示qPCR分析对预测局部和远端复发的灵敏度分别为42.3%和85.3%。与qPCR相比,加入cfDNA片段大小参数的EBV DNA靶向测序的灵敏度则分别为88.5%和97.1%,阴性预测值分别达到99.1%和99.4%。研究人员认为,基于EBV DNA靶向测序可以增强通过血浆EBV DNA标志物对鼻咽癌患者的预后评估[13]。
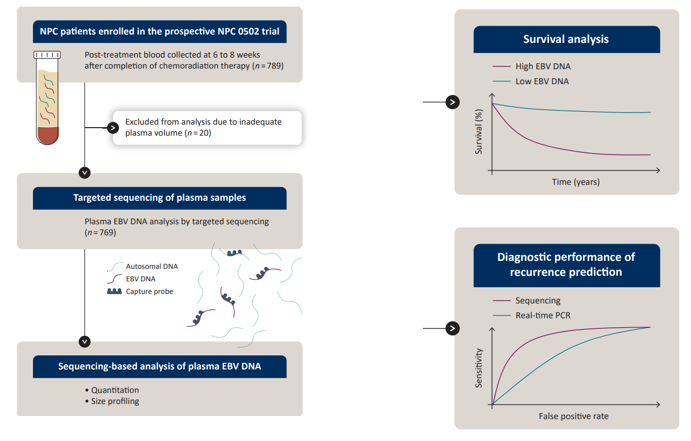
图7 加入cfDNA片段大小参数的EBV DNA靶向测序分析
上述cfDNA的片段大小特征都是基于双链DNA文库构建方法,有趣的是,最近的几项研究揭示了单链cfDNA分子在母体血浆cfDNA中的存在,尤其是在短DNA分子中[15、16]。然而,单链和双链DNA建库方法在评估胎源含量时似乎并没有差异[15]。此外,一些研究表明,使用改良的单链DNA文库制备方法研究发现,血浆cfDNA中存在大量以约50bp为中心片段(图7)。这些调整包括使用高亲和力磁珠(MB)提取cfDNA、基于Biotinylated Adapters的单链DNA文库制备方法[16],以及调整异丙醇的浓度以保留低分子量核酸[17]。值得注意的是,这种超短cfDNA片段被发现在血细胞的染色质可及性区域富集,特别是在可能含有G-四联体(G4)DNA二级结构的启动子区域[16]。据报道,这种富集现象在癌症患者中降低,可能为癌症检测提供了另一种类型的生物标志物[16]。
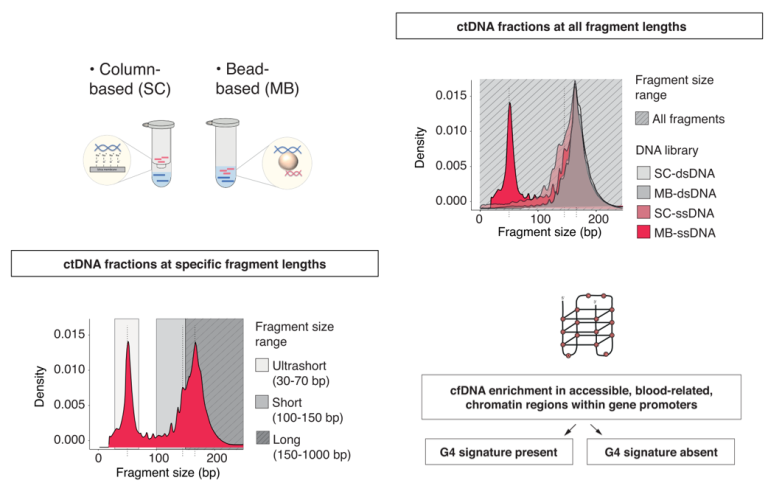
图8 不同提取和建库方法得到的cfDNA长度分布,并发现G4 DNA标志物
与大多数关注短cfDNA分子(<500 bp)的研究不同,Yu等人最近通过单分子实时(SMRT)长读长测序(Pacific Biosciences,PacBio)揭示了母体血浆中存在长链的胎儿cfDNA[18]。在母体DNA中检测到相当比例的长胎儿cfDNA分子,在妊娠的不同时期分别为15.5%、19.8%和32.3%,最长的胎儿cfDNA长度超过23kb。Yu等人进一步阐明,长cfDNA分子的比例可以作为先兆子痫的生物标志物。与短读长测序技术相比,通过长读长测序推断出的大小度量对有先兆子痫和无先兆子痫的妊娠之间有更好的区分[18]。
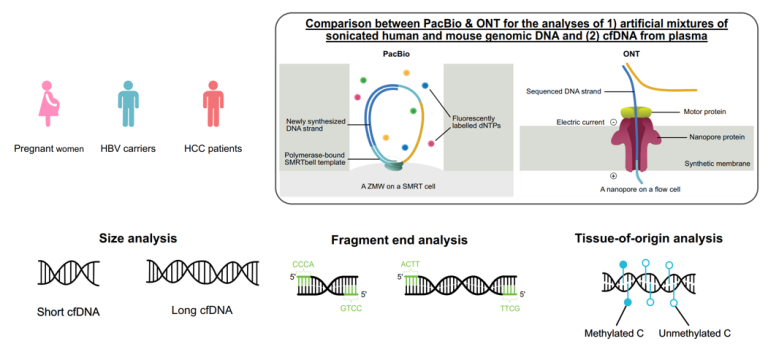
血浆中长链cfDNA的存在[18]可以将液体活检的边界从短cfDNA分子扩展到Kb级cfDNA分子。长链cfDNA的一个重要意义在于,使用一系列CpG的甲基化模式理论上可以确定每个cfDNA分子的组织来源。通过使用人工智能算法,Tse等人验证了使用长读长测序(SMRT)产生的动力学信号对天然DNA分子进行直接甲基化检测[整体动力学(HK)模型][19]。通过HK模型,可以凭借“固有分子条形码”追踪每个DNA分子的细胞来源。Yu等人在孕妇的母体血浆中证明了这一概念,利用长链cfDNA分子上CpG位点的丰富性,以0.88的准确率识别胎儿来源的DNA分子[18]。另一项独立研究表明,长读长测序能够对癌症患者进行长链cfDNA检测和直接甲基化分析[20]。这样一项概念验证研究表明,与AUC为0.75的短cfDNA相比,使用长链cfDNA分子可以明显提高检测性能,AUC达到0.91[20]。因此,以前未鉴定的长链cfDNA分子可能为NIPT和癌症检测中的液体活检创造许多新的可能性。