CAPLRseq,RNA靶向长读长测序辅助林奇综合征诊断
- boke
- 2023-03-28
- 7:07 上午
肿瘤抑制基因的DNA遗传变异检测可以识别具有肿瘤遗传易感性的个体。然而,超过44%的遗传变异通过DNA测序鉴定为临床意义未明的变异(VUS)。VUS不确性的影响可能阻碍患者及其家庭成员错过最佳预防或治疗时机,带来了临床上的巨大挑战。15%至25%的VUS会扰乱mRNA剪接,而遗传性肿瘤易感性基因极易受到剪接缺陷的影响 [1]。例如,在遗传性非息肉病性结直肠癌(HNPCC)/Lynch综合征的研究中发现,在错配修复蛋白MLH1、PMS2、MSH2和MSH6中,16%的错义突变和12%的VUS会造成剪接缺陷 [2]。
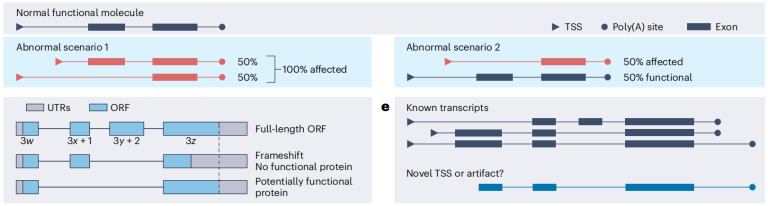
图1 不同变异带来的转录错误假设 [3]
长读长测序适用于检测由DNA变异产生的mRNA异构体。例如,有研究通过PCR扩增cDNA,再利用纳米孔测序技术(ONT)鉴定出32种BRCA1 mRNA的选择性剪接异构体,其中20种是新鉴定的 [4]。使用相同的方法,一项研究发现了NF1 mRNA的SNV变异和异常剪接体 [5]。然而,PCR技术通量较低,不适合多基因高通量检测和自动化。即使使用RNA-Seq通过ONT平台对整个转录组进行测序,但测序深度往往限制了低表达水平的新异构体的检测。

图2 BRCA1全长转录本测序 [4]
最近一些研究通过RNA靶向捕获技术进行深度长读长测序,可以灵活定制待检测靶基因,从头发现编码和非编码转录本。2023年1月2日Vincent Schwenk等人在《J Med Genet》上发表了题目为“Transcript capture and ultradeep long-read RNA sequencing (CAPLRseq) to diagnose HNPCC/Lynch syndrome”的研究,通过一种低投入量(50 ng)靶向富集系统(123 Gene Panel)与纳米孔技术相结合的方法,高效捕获遗传性肿瘤易感基因转录本。通过对疑似HNPCC/Lynch综合征患者样本的研究,证明了该技术可以解释变异对mRNA结构或表达的影响。
从全血或短期培养的外周血单核细胞中提取总细胞RNA。利用ONT cDNA-PCR测序试剂盒通过oligo-dT引物和模板反转录RNA,然后进行PCR barcoding扩增。扩增后的cDNA进行液相基因芯片靶向捕获,将捕获的cDNA通过PCR扩增,并在GridION仪器上进行ONT测序。
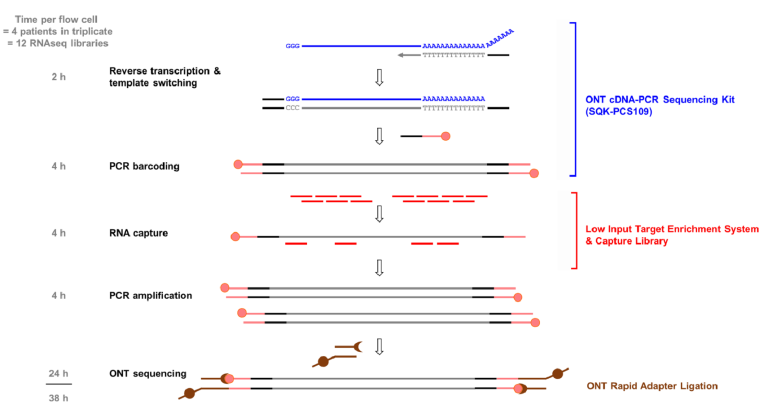
图3 RNA捕获和超深度长读长测序流程图
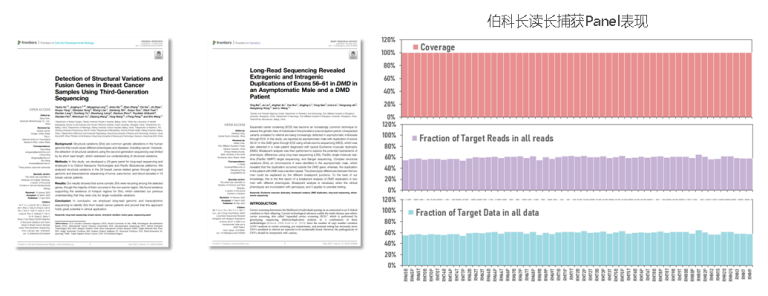
比较cDNA-seq或CAPLRseq富集后的123个遗传性肿瘤易感基因相关转录本的cDNA测序数据,结果显示94%的reads比对到参考基因组,最少也可以达到80%。cDNA-seq和CAPLRseq之间的读长没有任何系统性差异。并且,CAPLRseq方法将目标基因转录本的平均测序深度提高到5000x以上,提高超100倍。
通过证实MLH1、PMS2、MSH2和MSH6错配修复基因的剪接缺陷和等位基因表达缺失,该研究利用CAPLRseq确诊了17例HNPCC/Lynch综合征样本。此外,CAPLRseq还将MSH6和PMS2基因中两个意义不明的变异重新分类为可能致病或良性。
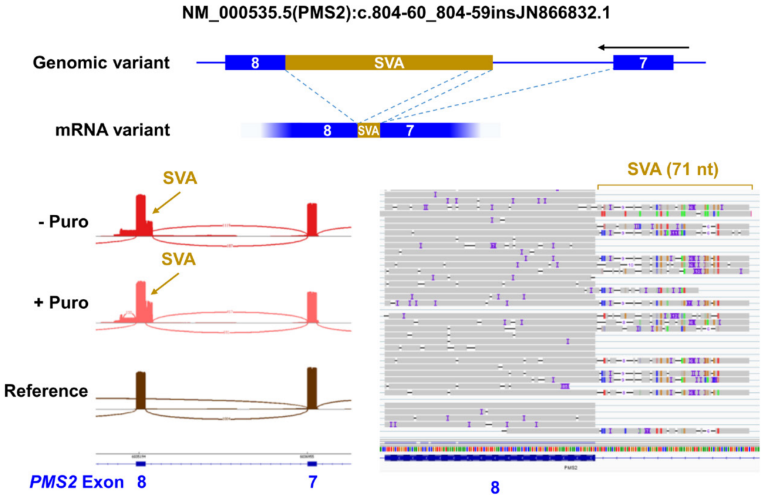
图4 CAPLRseq分析案例, 基因组层面,PMS2基因的7号内含子中存在SINE-VNTR-Alu(SVA)插入,mRNA检测发现8号外显子存在71nt的延伸
RNA靶向长读长测序方法提供了一个强大的诊断工具,其主要优势在于可以识别不同基因组变异对同源mRNA结构完整性和表达的影响。虽然单个剪接也可以通过短读长RNA-seq评估,但只有长读长测序才能在单分子水平上反映转录组的完整多样性,并且由结构变异引起的转录本改变通过短读长测序不易完成检测。RNA靶向长读长测序是一种高效的诊断方法,能够同时检测数个基因并实现自动化操作,具有成本效益,可以快速地整合到临床遗传学实验室的现有工作流程中。
伯科生物拥有靶向测序全流程国产化整体解决方案,已成功开展基于长读长靶向测序平台的遗传病、肿瘤和病原体检测等应用;伯科高品质核酸合成平台可以提供从探针设计和合成、个性化接头/Blocker设计生产、其他标准捕获配套试剂以及相关技术的开发优化服务,生产周期短、性能优异。