NGS最佳第六人- RNA-Cap
2021-11-15
融合基因检测对临床诊疗和预后均有着重要意义,融合位点常位于重复区域内,重复区域内的探针设计是捕获难题,比如某个基因的CNV Panel的设计,为了增加覆盖密度,加入重复区域探针后,中靶率从70降低至50%,而ROS1的Intron31添加至CDx Panel后则从中靶率从60降低至40%,Panel测序成本增加明显,因此,对于DNA-Cap Panel的融合热点的覆盖只能“忍痛割爱”,以牺牲融合检出为代价减少或者直接去掉相应的覆盖区域,再或者是通过反义DNA封闭off-Target区域,但是这些方法均有不足。
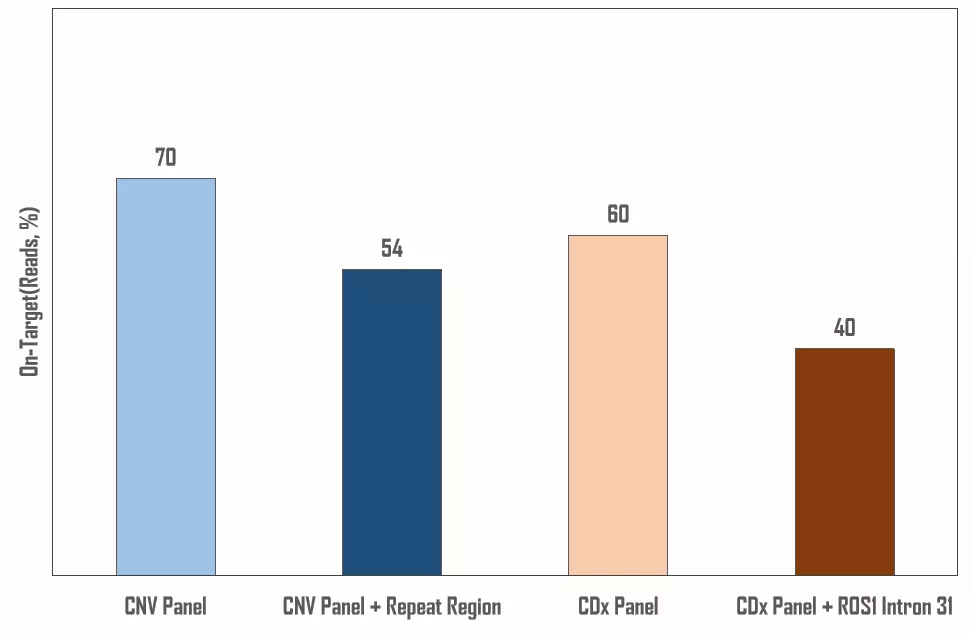
图1. DNA重复区对DNA-Cap捕获效率的影响
那么,避开DNA重复区,对RNA转录本进行检测就成了更优解。大量研究表明,RNA-Cap具有rRNA污染小,Exon区域占比高,起始量低,覆盖均一性好,耐受降解样本等诸多优势,明显优于mRNA-Seq和Ribo-Zero两种常用方法,可以说是各方面都更加适合复杂挑战样本的检测[1-3]。如今,RNA-Cap已经是DNA-Cap在融合基因和变异检测中的重要补充,例如FM的HEME One产品[4]。此外,添加ERCC外参、DNA污染监控模块使得RNA-Cap检测流程更加完善,但是否使得体系更加复杂不可控有待实践检验[5] 。
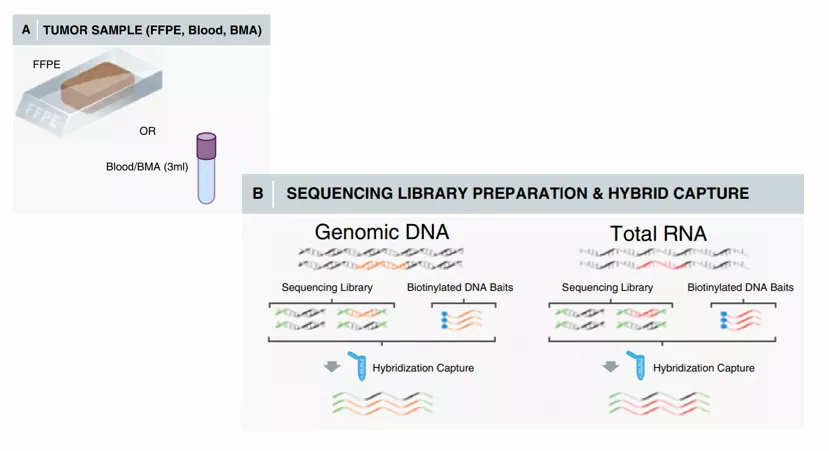
图2. HEME One,DNA+RNA-Cap[4]
随着肿瘤研究工作的积累,相应的伴随诊断需求日新月异,伯科生物拥有自主高通量合成平台,可在7个工作日内快速提供高品质ssDNA探针,助力科研、临床转化和产品升级迭代。以伯科自主合成的商品化RNA捕获Panel-Target Cap® Solid Tumor Fusion RNA-Cap Panel为例,该Panel包含95个实体瘤相关融合基因和3个内参基因,覆盖约200Kb编码区域。接下来,我们详细的介绍一下该Panel在FFPE融合基因标准品的融合基因检测中的表现。
实验设计
如图3所示,将FFPE标准品采用柱式提取法提取RNA,融合标品RNA RIN值较低(2.88),但DV200均较好(>70%)。取100ng Total RNA构建预文库(各3例),分别直接测序(Total RNA-Seq)和Target Cap® Solid Tumor Fusion RNA-Cap Panel捕获测序(RNA-Cap)后,对RNA-Cap Panel覆盖的约200Kb捕获区间进行统计分析。
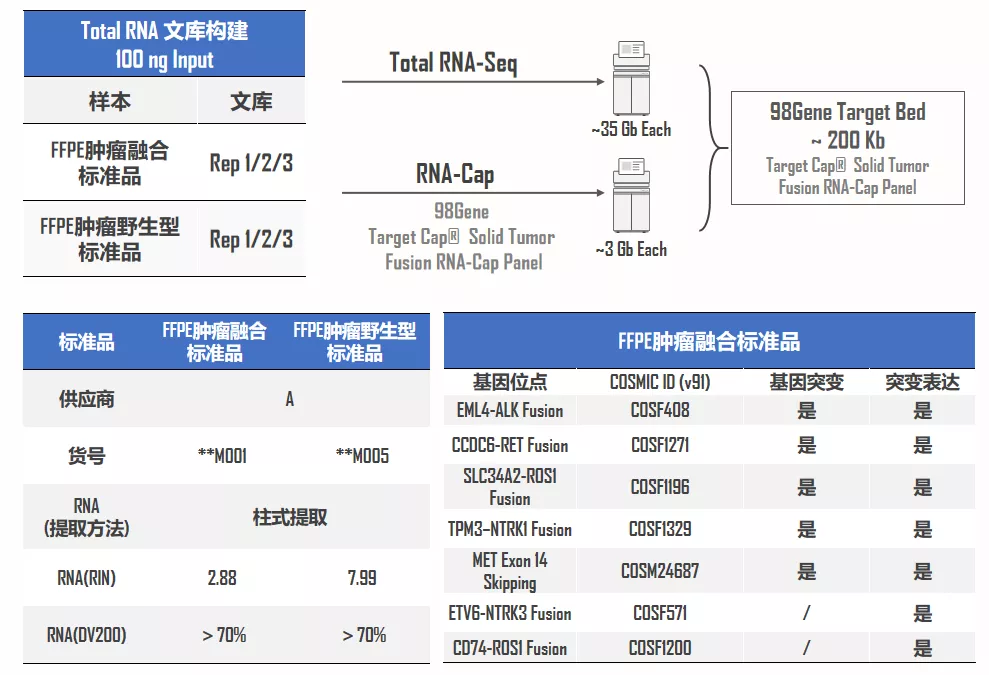
图3. Target Cap® Solid Tumor Fusion RNA-Cap Panel标准品测试
基本表现
如表1所示,RNA-Cap的On-Target为~70%左右,仅使用0.1Gb,RNA-Cap的测序深度已经达到200x左右,而Total RNA-Seq只有40~50x,富集效果明显。由于测序深度较高,RNA-Cap对转录本的覆盖率也明显优于Total RNA-Seq。

表1. Total RNA-Seq与Target Cap® Solid Tumor Fusion RNA-Cap Panel基本数据统计
Exon区域占比提升,是RNA-Cap数据的特征,与Total RNA-Seq ~40%的占比相比,RNA-Cap的Exon占比为~80%,将近提高一倍(图4上)。Total RNA-seq中的Intron区域数据多来自mRNA前体,伯科RNA-Cap针对转录本进行探针设计的策略,能够有效降低Intron区域占比,提高Coding区域数据有效性。此外,融合基因检出率依赖转录本覆盖均一性[6,7],伯科Target Cap® Solid Tumor Fusion RNA-Cap Panel在保持高效富集的同时,对基因转录本的覆盖均一性也同样优秀(图4下)。
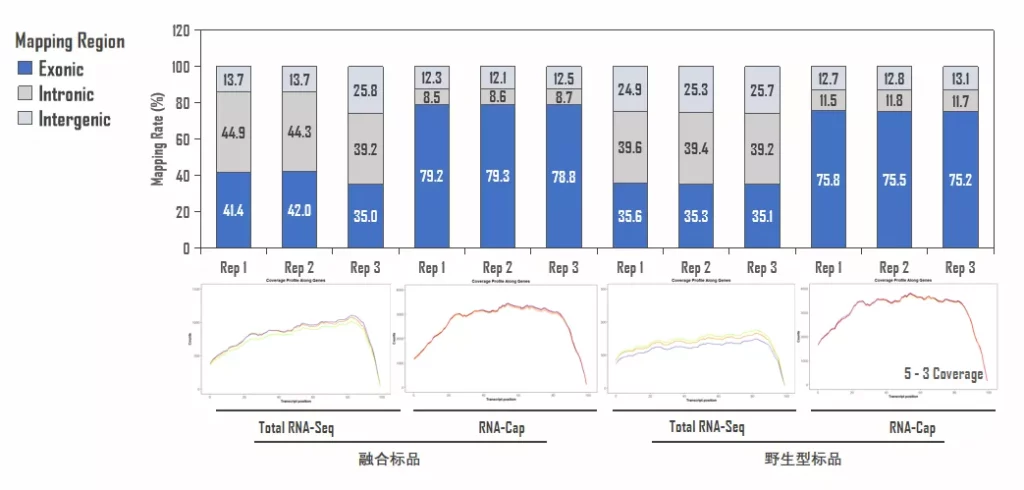
图4. Total RNA-Seq与RNA-Cap数据中基因组区域分布及转录本覆盖表现
融合基因检测
使用StarFusion软件对RNA-Cap以及Total RNA-Seq数据进行统计分析,如图5左所示,融合标品携带的7种融合变异,RNA-Cap全部检出,Total RNA-Seq则有3种未检出;同时,野生型标品均未检出。总的来说,RNA-Cap展示出极佳的灵敏度和特异性。此外,我们也观测到经典的融合表达上升的现象(图5右)。
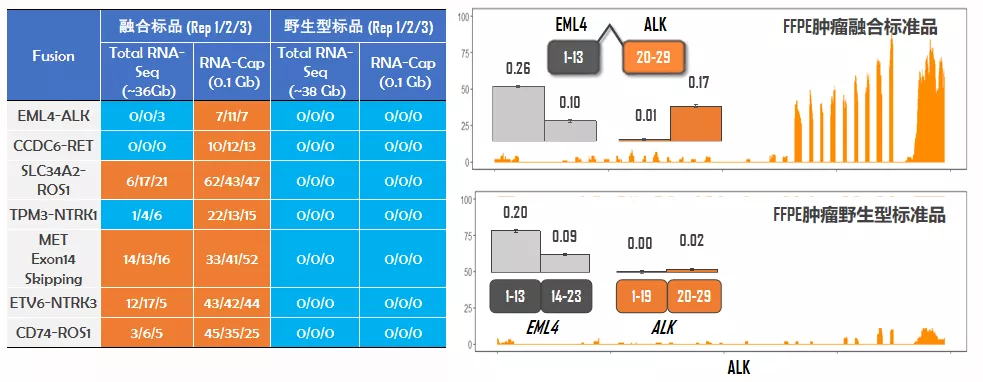
图5. 标准品融合基因检出及不同标品中ALK转录本覆盖
虽然RNA-Cap进行了高效的富集,但并没有产生明显的偏好,RNA-Cap的基因相对表达与Total RNA-Seq (~100 Gb)高度相关,R2>0.99。
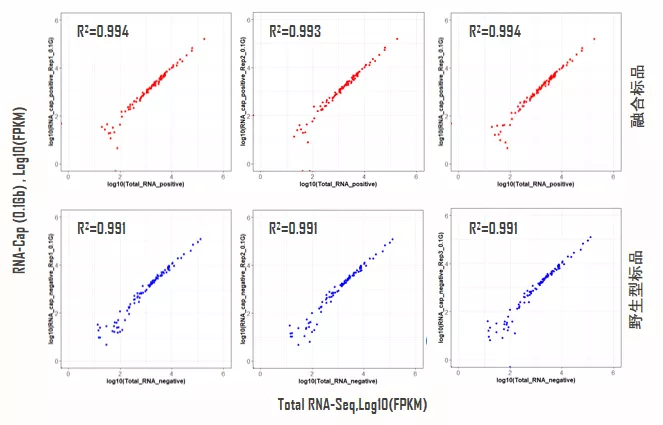
图6. 0.1Gb RNA-Cap与Total RNA-Seq基因相对表达量相关性
万变不离其“中”
融合基因伴侣的数量多、融合位点复杂多变,RNA-Cap的另一大优势是通过捕获骨架基因,检测多种融合伴侣和亚型。值得一提的是,在本次实验中,RNA-Cap Panel只针对ABL1设计了探针,并未涉及BCR基因,但在两种标品中,虽然BCR-ABL1融合变异并未纳入标品变异List,但RNA-Cap仍然发现了BCR-ABL1融合,提示FFPE标品的遗传背景中可能具有BCR-ABL1融合变异。除此之外,在其他阳性样本中,RNA-Cap也能够发现BCR-ABL1融合的不同亚型(图7)
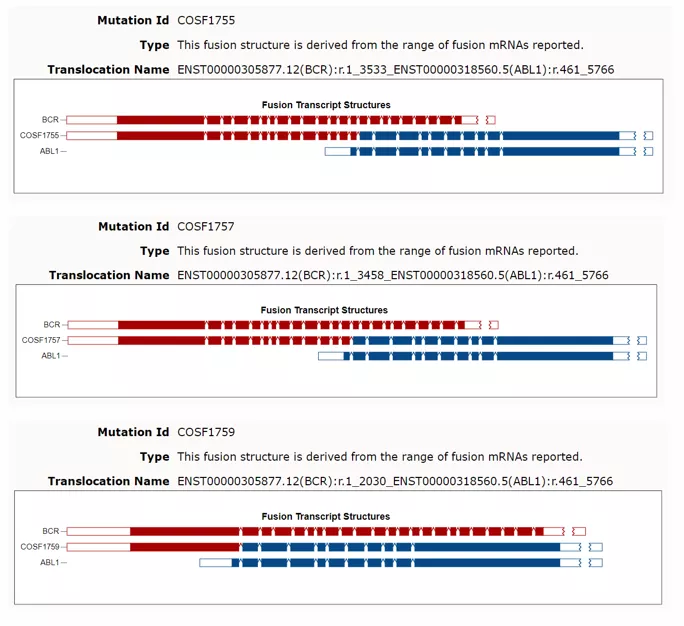
图7. 在不同阳性样本中,RNA-Cap检测到的部分BCR-ABL1 融合亚型
结语
通过近年的应用实践,RNA-Cap已经被证明是DNA-Cap的有效补充,其成本低,准确性高,能一次完成多个融合基因检测,同时耐受低质量样本等技术优势,使之与临床需求完美匹配。凭借过硬的技术参数,伯科RNA-Cap Panel广受好评;同时,伯科依托自主高通量合成优势,为用户产品迭代,技术创新提供了强大助力,例如开发DNA/RNA共捕获等创新检测技术。我们相信,RNA-Cap的应用将不仅限于融合基因的检测,伯科愿与同仁一道,进一步挖掘RNA-Cap技术的应用潜力。
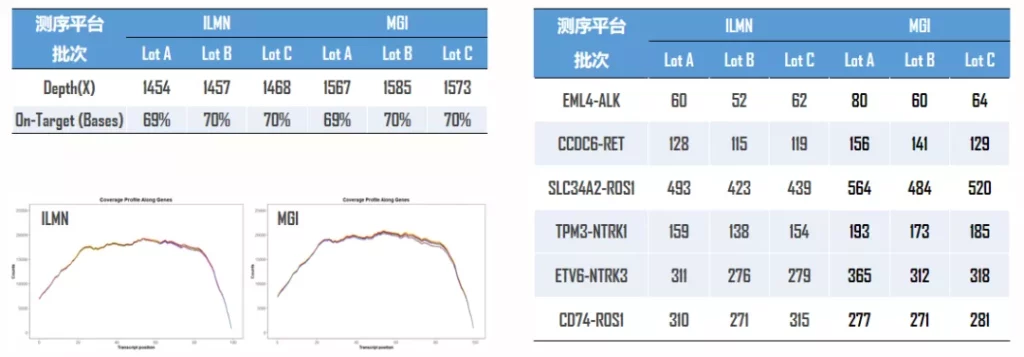
图8. Target Cap® Solid Tumor Fusion RNA-Cap Panel不同批次数据表现(1Gb)
参考文献:
[1]. Mercer, T. R. et al. Targeted sequencing for gene discovery and quantification using RNA CaptureSeq.Nat. Protoc. 9, 989–1009 (2014).
[2] Cieslik M , Chugh R , Wu Y M , et al. The use of exome capture RNA-seq for highly degraded RNA with application to clinical cancer sequencing[J]. Genome Research, 2015, 25(9):1372.),
[3]. Schuierer S, et al. A comprehensive assessment of RNA-seq protocols for degraded and low-quantity samples. BMC Genomics. 2017 Jun 5;18(1):442.\
[4] He J , Abdel-Wahab O , Nahas M K , et al. Integrated genomic DNA/RNA profiling of hematologic malignancies in the clinical setting[J]. Blood, 2016, 127(24):3004.
[5] Reeser J W , Martin D , Miya J , et al. Validation of a Targeted RNA Sequencing Assay for Kinase Fusion Detection in Solid Tumors[J]. Journal of Molecular Diagnostics Jmd, 2017.
[6]. Gocke, C. D. et al. Risk-based classification of leukemia by cytogenetic and multiplex molecular methods: results from a multicenter validation study.Blood Cancer J. 2, e78 (2012). [7] Davila, Jaime I. , et al. Impact of RNA degradation on fusion detection by RNA-seq. BMC Genomics 17.1(2016)