伯科TAPS-Cap助力ctDNA表观研究
异常的DNA甲基化与各种疾病有关,是公认的癌症特征。cfDNA从细胞中释放后,依然保持着部分生物学特征,包括基因组和表观组两类重要信息。在表观特征中,cfDNA甲基化修饰不但具有定位肿瘤的潜质,还能够从组学层面增加ctDNA标志物检测的“广度”,提高ctDNA检测灵敏度。cfDNA甲基化标志物已被广泛应用于癌症早期筛查、复发监测等领域的研究。
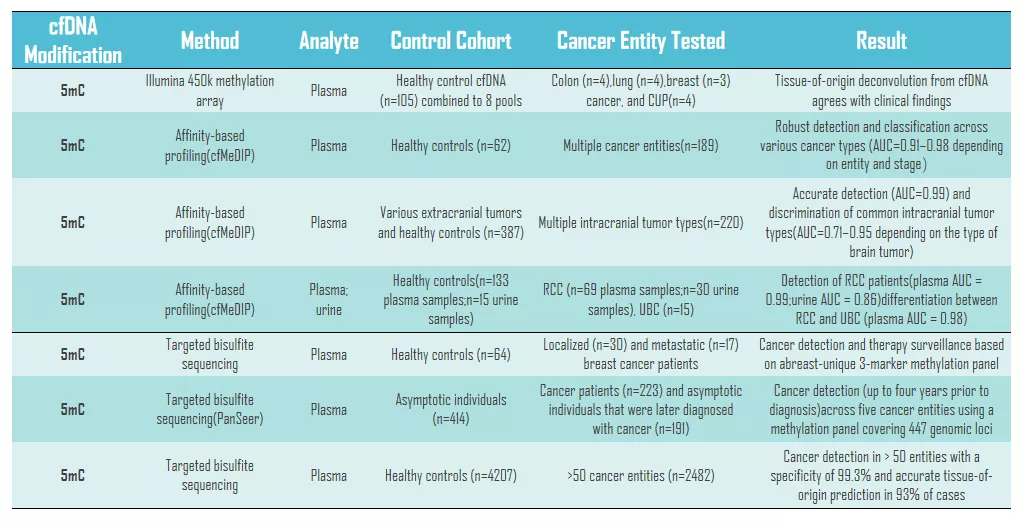
表1. 基于cfDNA甲基化标志物的癌症无创检测研究[1]
NGS技术出现后,基于亚硫酸氢盐(Bisulfite)的全基因组甲基化测序(WGBS)将甲基化的分辨率提升至单碱基水平,研究人员可以获得更为精确的甲基化图谱,为ctDNA的溯源定位,提供更加准确的“指纹”信息;但Bisulfite是一把双刃剑,Bisulfite处理后序列丰富度降低,DNA单链化,非随机片段化,WGBS比对率低,覆盖不均匀、冗余高,整体数据有效性较低;单链建库技术的进步在一定程度上缓解了这一问题,但缺乏温和高效的转化技术仍然制约着cfDNA甲基化标志物的研究发展。
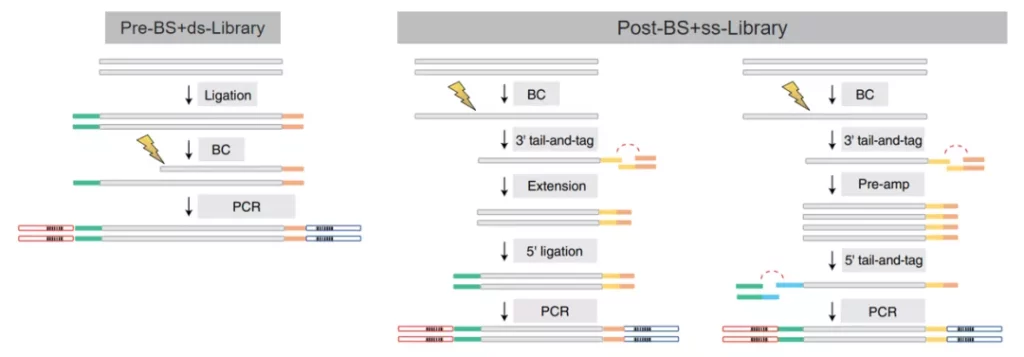
图1.BS建库技术进展[4]
EM-Seq、TAPS等BS-free转化技术的出现,正在逐渐改变这一局面。EM-Seq利用TET/BGT保护5mC碱基避免APOBEC酶的转化,TAPS则是将5mC转化为可被吡啶硼烷(Pyridine Borane)“识别”的5caC,C和5mC在转化后最终形成不同碱基,前者是纯酶学方法,后者是半酶学半化学方法。
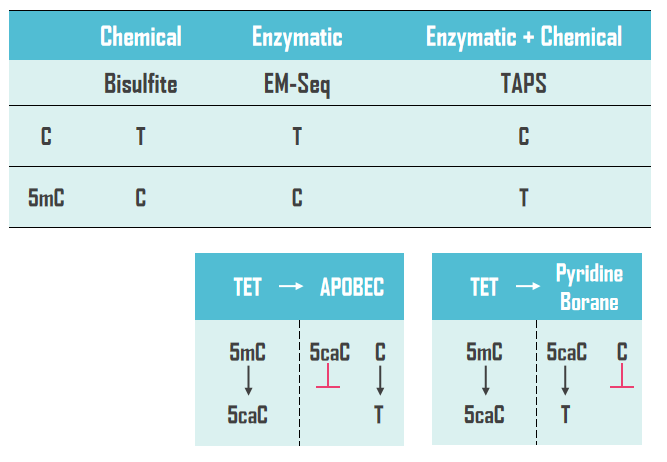
图2. BS-free技术原理简化示意图[2]
BS-free技术的低损伤优势可以让DNA保留更多的原始信息,比如cfDNA的片段化特征(图3),其中,TAPS测序还能够保持序列丰富度,有利于下游分子操作和测序,比如引物和探针设计,同时测序的碱基均衡性更好。因此,BS-free技术在表观特征研究以及多组学标志物联合检测中拥有巨大潜力。
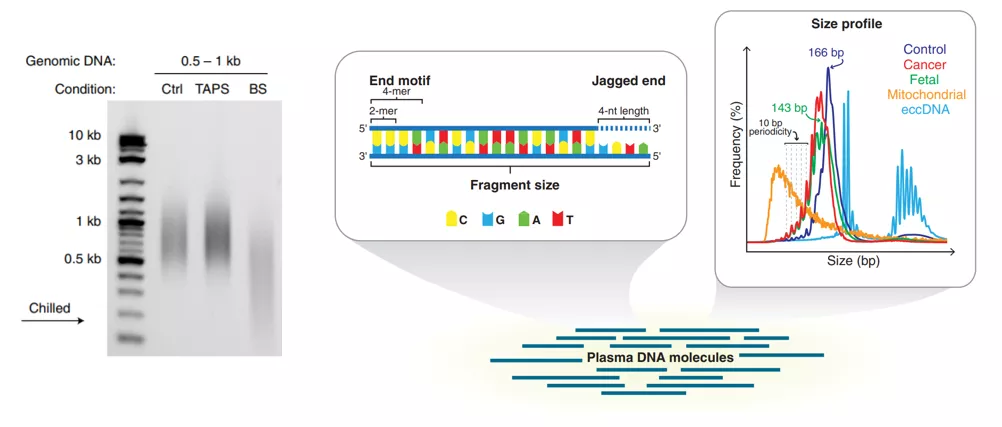
图3. TAPS的“无伤”优势有利于cfDNA表观特征研究[2, 3]
伯科TargetCap捕获技术针对BS-Seq、EM-Seq和TAPS均有成熟的靶向测序解决方案;需要注意的是,如图4左所示,由于只转化5mC而且甲基化Panel主要集中在CpG区域,TAPS-Cap Panel的探针GC更高,易形成二级结构,针对TAPS-Cap探针生产,伯科配套特殊的制备工艺,保证TAPS探针质量(图4)。
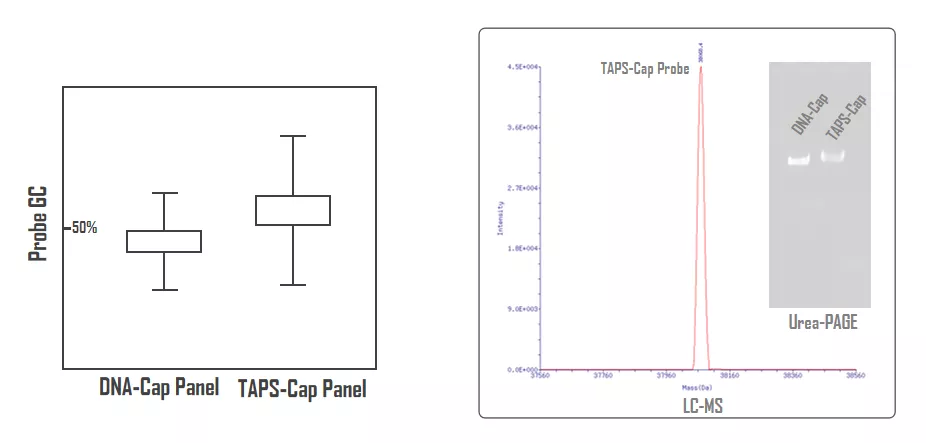
图4. DNA-Cap与TAPS-Cap Panel探针比较
使用TAPS-Cap Panel与DNA-Cap Panel对非TAPS处理的Coriell gDNA预文库(模拟低甲基化样本)进行捕获,TAPS-Cap Panel的基本表现,覆盖率、中靶率、覆盖均一性与DNA-Cap接近,同时批次稳定(图5)。

图5. 伯科DNA-Cap和TAPS-Cap Panel对非TAPS处理DNA的捕获表现
对DNA进行TAPS处理,随后采用伯科和友商I**的相同目标区域TAPS Panel进行杂交捕获。两款Panel对基因组的覆盖高度相关,R2=0.96,同时,捕获性能和甲基化分析比较也显示两款Panel性能相近(未展示),表明伯科TAPS-Cap Panel已能比肩国际竞品。
相信随着工程酶和工艺流程的不断优化,BS-free技术还有很大提升空间。我们也期待研究人员不断给我们带来惊喜,持续推进相关领域的发展。成立以来,伯科在核酸合成和靶向捕获领域积极开拓,积累了丰富的项目经验,依托自主合成平台,伯科能够快速响应用户需求,提供切实可行的解决方案,我们也希望在更多的领域中提供底层技术助力。
参考文献:
1. Angeles A K, Janke F, Bauer S, et al. Liquid Biopsies beyond Mutation Calling: Genomic and Epigenomic Features of Cell-Free DNA in Cancer[J]. Cancers, 2021, 13(22): 5615.
2. Liu Y, Siejka-Zielińska P, Velikova G, et al. Bisulfite-free direct detection of 5-methylcytosine and 5-hydroxymethylcytosine at base resolution[J]. Nature biotechnology, 2019, 37(4): 424-429.
3. Lo Y M D, Han D S C, Jiang P, et al. Epigenetics, fragmentomics, and topology of cell-free DNA in liquid biopsies[J]. Science, 2021, 372(6538).
4. Haque I S. Enhanced DNA libraries for methylation analysis[J]. Nature Biomedical Engineering, 2021: 1-3.


