迈向融合基因检测2.0时代——伯科合成助力肿瘤融合基因精准检测
2019-12-07
以下文章来源于测序中国 ,作者 伯科生物

染色体重排所导致的基因融合是肿瘤中的常见现象,在肿瘤的发生过程中起着非常重要的作用,约占人类癌症发病率的20%。目前,国内外已有多种抑制融合基因的肿瘤靶向治疗药物用于临床,包括伊马替尼/BCR-ABL1、克唑替尼/EML4-ALK和拉罗替尼/NTRK融合等。
然而,融合基因发生的频率在不同的癌症中存在很大的差异,许多融合基因只存在于特定的癌症亚型。例如,非小细胞肺癌(NSCLC)中ALK融合基因阳性率为3%~7%;仅2%的ROS1融合基因存在于肺癌患者中;所有实体瘤中NTRK基因融合阳性率平均约为1%,而在某些特定肿瘤中却高达90%[1,2]。
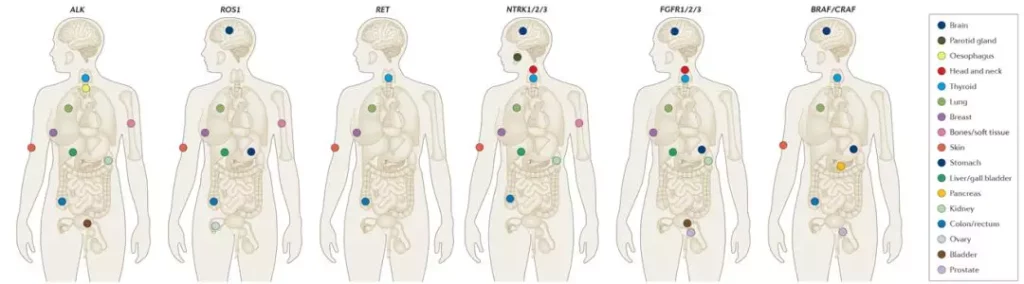
机遇与挑战丨融合基因精准检测如何实现?
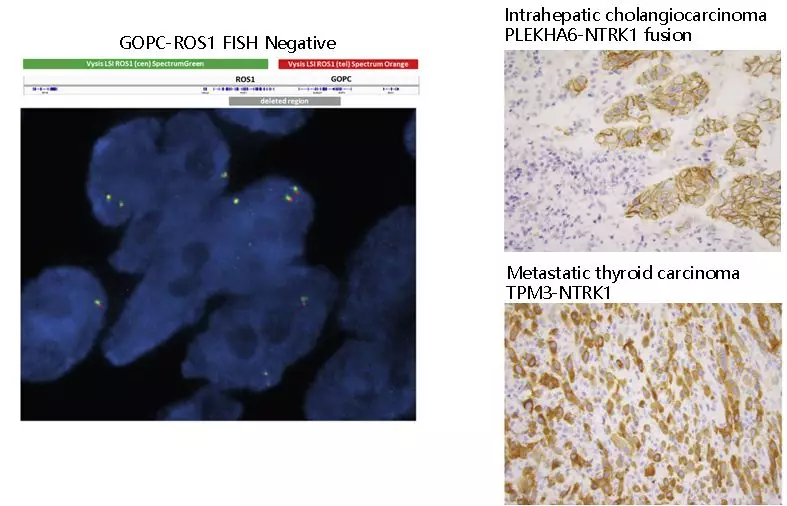
因此,精确检测融合基因不但可以协助临床医生对癌症进行诊断和分型,还能够为后续的治疗提供必要信息。目前,融合基因诊断主要包括荧光原位杂交(FISH)、IHC等。然而,这些检测方法的分辨率和通量通常较低,同时还较为依赖检验人员的判断经验,而且往往只适用于已知的融合亚型,应用拓展有限,因而常常导致诊断过程漫长、费用昂贵。如图2所示,由于Deletion区域较小,导致探针仍然可以与目标区域结合,因此,FISH结果并未检测到GOPC-ROS1融合,出现了假阴性的情况。有报道表明,使用IHC检测NTRK3融合的灵敏度降低,其中,NTRK1和NTRK2的灵敏度分别为97%和99%,而NTRK3只有79%;此外,其特异性还与肿瘤类型相关。[5]
尽管传统检测方法仍将作为肿瘤融合基因诊断的不可或缺的手段,但与此同时,多种新型技术平台也在逐步走进临床(表1)。例如,基于杂交/荧光或者质谱技术的融合基因检测手段在一定程度上避免了技术人员的主观判断,更加数字化,在通量提高的同时还具有经济优势;但另一方面,现阶段临床研究已发现的肿瘤融合基因超过了10000种,虽然以上技术都具有较高的检测灵敏度,但它们都不能发现新的融合亚型,仍存在一定局限性。除此之外,基于NGS的锚定扩增子技术虽然方便快捷,也不需要融合的先验知识,但稍显不足的是容易产生假阳性,需要利用生物信息管线过滤掉置信度低的融合基因。
表1. 新型基因融合检测方法[4]

多维对比丨RNA-Cap改善临床融合基因检测潜力巨大
值得关注的是,Hybridization-based DNA靶向测序技术(DNA-Cap)近年来凭借其经济、不依赖融合先验知识、新融合亚型的发现能力,以及适用于基因组和游离核酸的广泛性、高灵敏度等诸多优势,越来越多地被广大临床检验实验室所采用。
当前市面上,多款基于NGS技术的DNA-Cap伴随诊断产品都具有检测融合基因的能力,例如MSK-IMPACT等。其整体流程如下:设计覆盖功能基因内含子区域的探针,将探针与预文库杂交,捕获测序,分析统计嵌合型Reads,从而发现融合事件。但单独使用DNA-Cap检测融合基因也有一定的局限性:
1. 灵敏度
NGS技术的灵敏度与覆盖度直接相关,有些内含子非常长,如NTRK3中的内含子 (~193kb)。如果覆盖,将进一步增加成本。此外,有些内含子区域即使在需要的情况下也无法设计探针,因为它们包含了大量重复序列,会降低on-target,同时也较难比对。例如,ROS1的31号内含子,作为主要融合区域(CD74/SLC34A2/SDC),含有90%以上的重复序列。因此,如果融合断点涉及未覆盖的内含子区域,而捕获探针无法覆盖这些区域,检测灵敏度将受到影响。例如, 在某些FDA批准的肿瘤大Panel中,对于NTRK融合通常采用热点内含子部分覆盖和伴侣覆盖(NTRK3-ETV6),可能有漏检的风险(图3)。
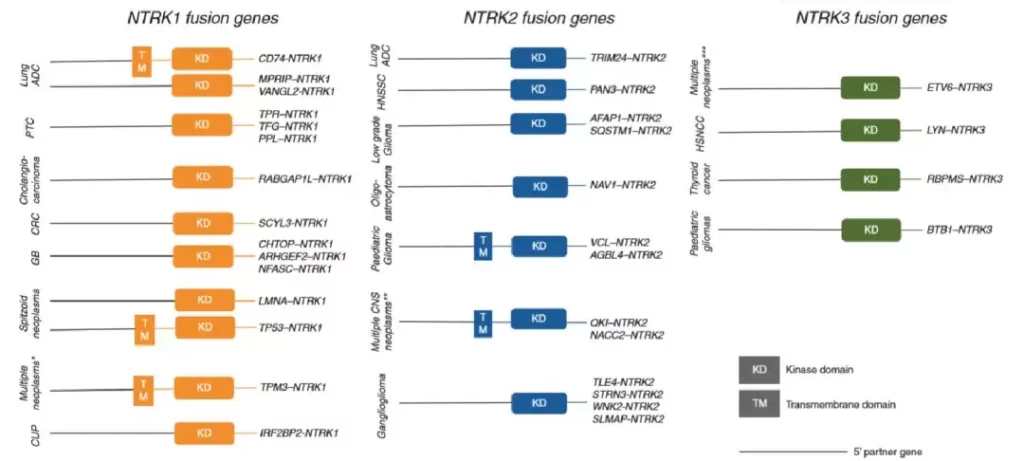
2. 特异性
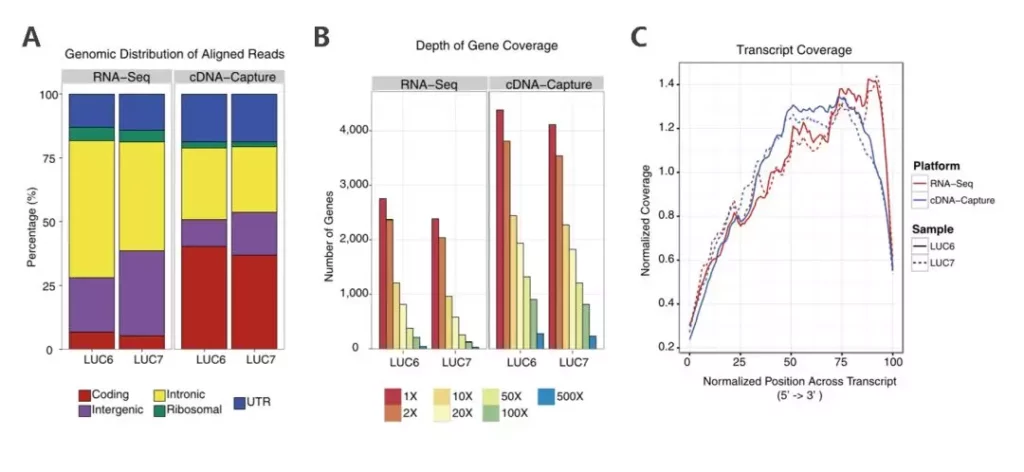
不同的分析软件导致的假阳性率不同,因此,有研究同时采用多种融合分析软件以提高特异性[7];更加值得注意的是,基因组层面的染色体重排或基因融合很难确认融合基因是否表达或功能性。在一项基于NGS的DNA/RNA研究中,使用MSK-IMPACT Panel发现的23例NTRK融合阳性样本,其中21例确认具有融合转录本表达,两例阴性样本既没有融合转录本表达(Archer RNA testing)也未检测到融合蛋白(IHC),该研究显示了对基因组融合进行验证的重要性[8]。尽管有一些研究使用全转录组测序来检测融合基因,但是由于全转录组测序在病理检测后,肿瘤临床样本多以FFPE为主的,其RNA一般都有部分降解,质量较低。基于NGS的RNA-seq技术中,mRNA-seq局限性较大,在一项使用mRNA-seq对乳腺癌融合基因的研究中,只有45.7%(32/70)的融合基因可以被RT-PCR和Sanger方法验证[9];而使用去核糖体方法时,由于mRNA前体的存在,Intron含量增加、Exon占比较低,由于RNA降解、去rRNA不充分,表现为有效数据低且不稳定(1%-30% Exon ratio);除此之外,组织中的非目标基因还将进一步压缩目标Exon的数据占比。换言之,全转录组测序可以被理解为天然的“WES”,但是由于基因的差异化表达,转录本覆盖会参差不齐,同时又限于实验技术(去核糖体、逆转录等)和生物学(mRNA前体)方面的影响,最终可能需要大量的数据量来提升RNA-seq对融合基因的检测灵敏度和稳定性(图4)。
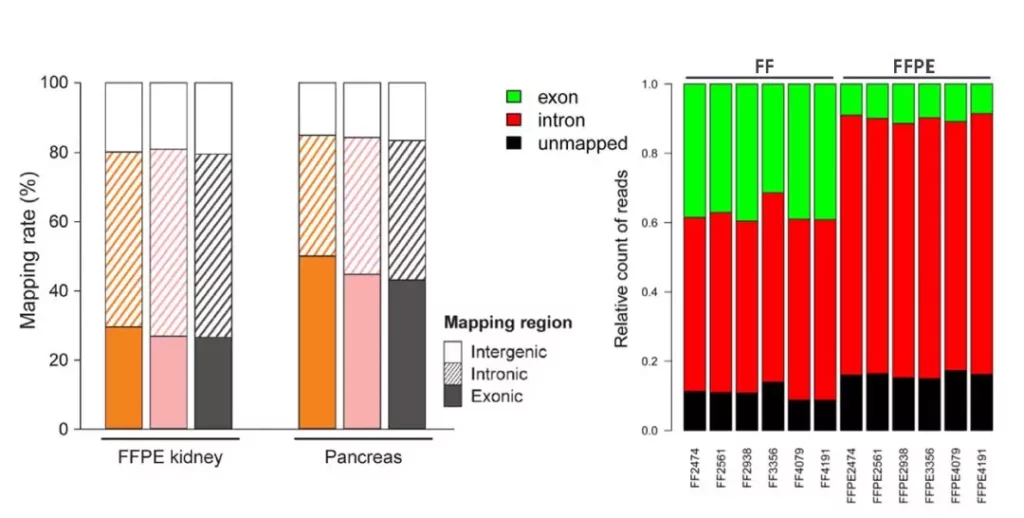
相较而言,RNA-Cap技术在融合基因检测方面则可以克服上述诸多瓶颈。首先,针对转录本设计探针避免了内含子区域的干扰,有效地提高了灵敏度且Panel更小、更加经济;其次,有效规避rRNA污染以及内含子和非目标基因干扰,提高数据有效性和目标区域深度,增加检测灵敏度(图5);更为重要的是,RNA-Cap还能够在发现融合基因的同时对融合基因的功能(in-frame)和表达量进行评估,对融合基因进行二次验证。正因如此,现在MSK-IMPACT对入选患者使用DNA-Cap和RNA锚定扩增子技术进行共检测。
未来可期丨本土化液相芯片杂交捕获技术助力融合基因检测
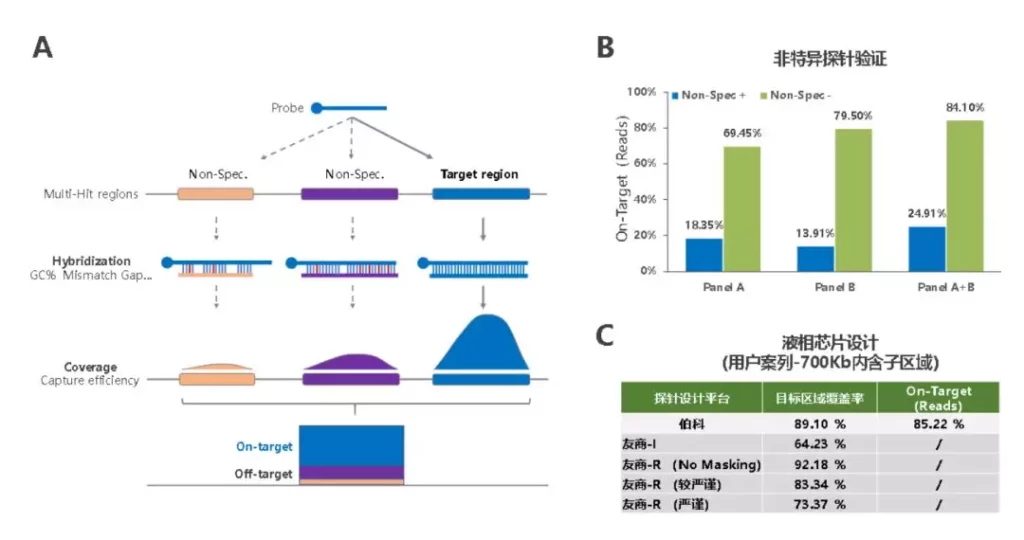
随着测序成本的不断降低、NGS测序仪的本土化,以及肿瘤临床应用在国内获批进入临床,DNA Panel可以进一步提高内含子区域的覆盖来增加肿瘤融合基因的检测灵敏度,而内含子区域往往存在大量的重复序列,探针设计困难重重。那么,如何达到覆盖率与On-target的平衡呢?国内自主知识产权的液相芯片杂交捕获技术供应商伯科生物近年来通过“实验-学习-验证”的系统摸索,建立了探针特异性(长度、GC、错配和Gap等参数)与捕获效率的数学模型,形成了其独有的Panel On-target预测系统(图6A)。在一个项目中,该公司用户直接提供探针区域用于合成,其产品研发团队在预测On-target并与用户沟通后,删除了不涉及临床热点突变的~0.5%的“Bad”探针,验证实验表明,在删除“Bad”探针后,Panel A(170Kb)和Panel B(230Kb)的On-target从18.4%和13.9%,提升至69.5%与79.5%,满足产品预期(图6B)。在另一款定制产品中,针对用户提供的~700Kb的内含子区域,该公司探针设计覆盖率达到89%,远高于友商I的64%,同时,On-target达到85%以上,表现优异(图6C)。
对于RNA-Cap Panel,伯科生物针对全长转录本设计探针,并对多转录本和可变剪接特殊设计,同时提供DNA污染监控模块、ERCC外参探针模块以及常见融合型探针供用户选择。据悉,目前伯科生物已完成多款RNA-Cap的设计与合成,并助力临床需求对DNA/RNA共捕获技术的开发。在将DNA-Cap和RNA-Cap整合为共捕获流程后,操作简便,可以对SNV、Indel、Fusion等多种突变类型进行检测,同时基因组与转录组相互验证,结果更加准确;此外,还可以进行组织溯源和分子分型。如图7所示,DNA&RNA共捕获流程较单独使用DNA-Cap时,融合基因检测灵敏度更高,此外,在基因表达量分析方面,该流程与RNA-seq也具有极高的相关性。

结 语
同时评估基因组变异与变异基因表达,通过DNA&RNA共捕获对融合基因的二维检测方式,可以说是精准医疗发展的一个缩影,从一药一检到肿瘤小Panel/大Panel,再到WES,肿瘤伴随诊断技术正以前所未有的速度不断完善,相信随着技术的成熟和测序成本的降低,高通量测序必将更好的服务于患者。伯科生物拥有国内独家的高通量核酸合成与修饰技术,致力于建立从寡核苷酸、DNA/RNA序列片段到染色体基因组成和构建的全流程合成生物学平台,目前该公司已经完成多组学基因芯片的设计软件、合成平台以及高性能杂交缓冲液和Blocker产品的开发,形成了针对不同应用场景的多种整体解决方案,并为大量用户提供了包括甲基化、Nanopore纳米孔靶向测序在内的基因芯片产品。基因Panel的设计与合成等上游技术此前一直被国外公司垄断,如今,随着以伯科生物技术为代表的具有我国自主知识产权的技术平台与产品问世,相信它们在未来将创造更大价值,为中国精准医疗事业的发展积极助力。
Website:www.bokebiotech.com
参考文献
1. Expression of the ETV6-NTRK3 gene fusion as a primary event in human secretory breast carcinoma.
2. Mammary analogue secretory carcinoma of salivary glands, containing the ETV6-NTRK3 fusion gene: a hitherto undescribed salivary gland tumor entity.
3. Fusions in solid tumours: diagnostic strategies, targeted therapy, and acquired resistance.
4. Overview of Fusion Detection Strategies Using Next-Generation Sequencing.
5. Identifying patients with NTRK fusion cancer.
6. ESMO recommendations on the standard methods to detect NTRK fusions in daily practice and clinical research.
7. Diagnosis of fusion genes using targeted RNA sequencing.
8. Pan-Trk immunohistochemistry is an efficient and reliable screen for the detection of NTRK fusions.
9. Recurrent Fusion Transcripts Detected by Whole-Transcriptome Sequencing of 120 Primary Breast Cancer Sample
10. Comprehensive comparative analysis of RNA sequencing methods for degraded or low input samples
11. cDNA Hybrid Capture Improves Transcriptome Analysis on Low-Input and Archived Samples
12. Robust gene expression and mutation analyses of RNA sequencing of formalin-fixed diagnostic tumor samples





