用户测评|伯科生物全外显子芯片v3.0(一)
2021-09-22
随着生命科学与生物技术的快速发展,藏在基因序列的秘密正在被逐步探索。全基因组重测序能挖掘到样本在全基因组范围的大部分遗传变异,但非编码区约占全基因组区域的70-80%,外显子捕获测序能够帮助研究人员将注意力与资源集中在基因组编码区。尽管有一些基因的调控因子位于非编码区,但综合考虑数据测序成本、数据存储成本、数据分析时长等因素,全外显子捕获后测序仍是目前最具性价比的测序手段。本篇文章旨在展示伯科生物TargetCap® Core Exome Panel v3.0与友商I** v2数据表现。
测评简介
01 全外芯片
伯科生物TargetCap® Core Exome Panel v3.0及友商I** v2
02 测评过程
通过平行干、湿实验,比较两款全外芯片性能
03 测评单位
所有实验均由第三方机构独立完成
04 测评方案
测试样本采用gDNA,杂交方式为多样本混合、过夜杂交
05 测序平台
MGI(DNBseq)华大智造测序平台
实验设计
本次测评实验共通过9组(9 x 2=18)样本进行对照实验,采用多样本混合(>3-Plex),过夜杂交的方式进行外显子区域捕获,并通过MGI测序平台进行测序。两款全外显子芯片捕获区域大小相近,伯科生物TargetCap® Core Exome Panel v3.0(下文简称“伯科v3.0”) 和友商I** v2 分别为42Mb和39Mb;值得注意的是,上述两款芯片设计分别采用“智能映射”和“捕获感知”算法,他们的表现究竟如何呢?
图1. 测评实验流程图
测评结果
两款全外显子芯片分别得到9组测序数据,除友商I** v2 8#样本测序深度在100X 以下,其余17份样本测序深度均大于100X,并且比对率、捕获文库Insert均一致。此外,除1#和4#样本外,其余样本两款芯片测序深度差异较大,在覆盖均一性比较中主要关注1#和4#样本。
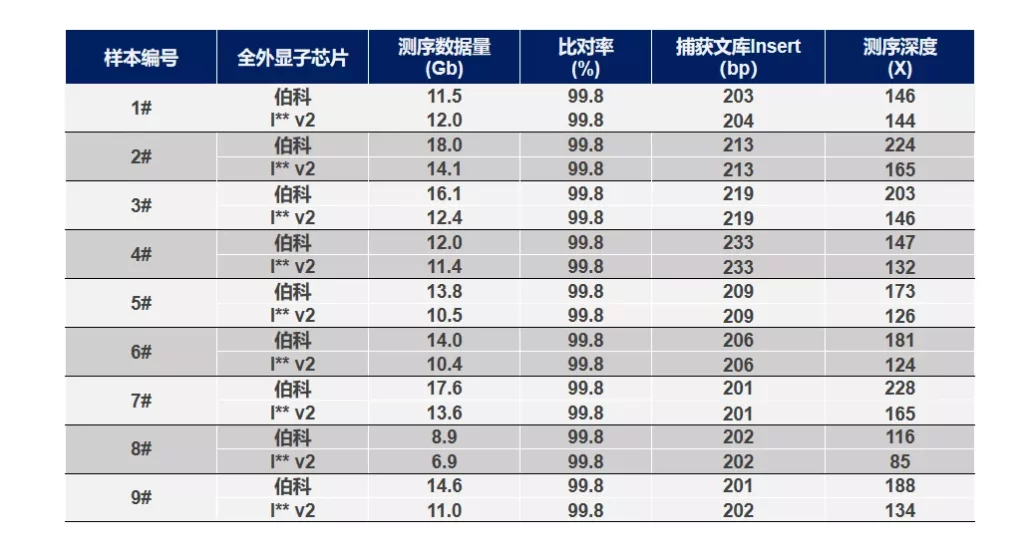
图2. 基本测序信息
接下来,我们主要从以下几个方面测评全外芯片的捕获表现:
1. 冗余度:测序数据尽可能低的冗余;
2. 中靶率:测序数据尽可能多位于目标区域内;
3. 目标区域覆盖率:能否将目标区域尽可能完整的覆盖;
4. 数据有效性:测序数据尽可能少浪费(每Gb测序深度)。
如图3所示,两款全外芯片表现如下:
1. 冗余度:伯科v3.0低于友商I** v2;
2. 中靶率:伯科v3.0持平于友商I** v2;
3. 目标区域覆盖率:伯科v3.0持平于友商I** v2;
4. 数据有效性:伯科v3.0略优于友商I** v2。
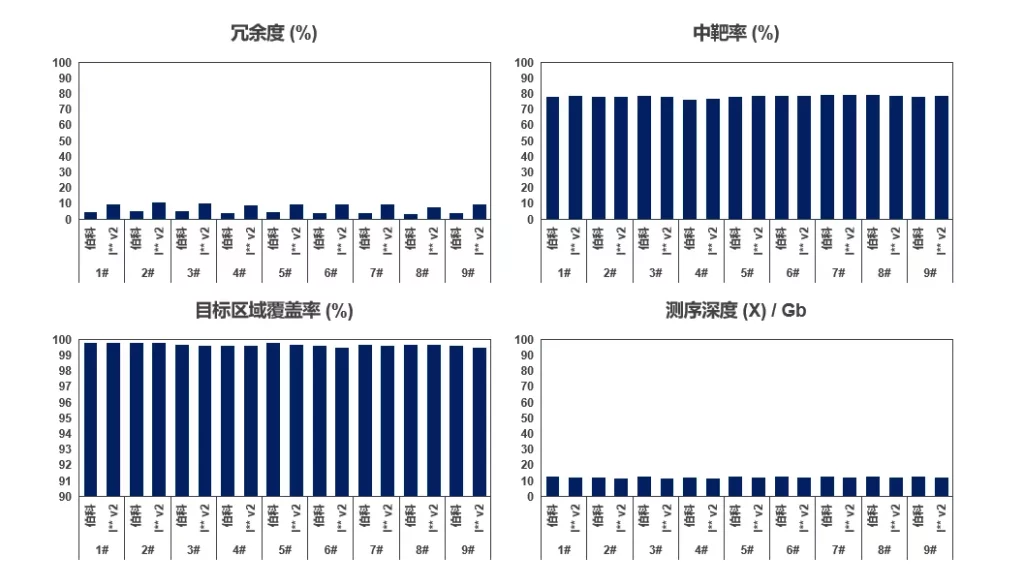
图3. 全外显子芯片捕获表现
(冗余度/中靶率/目标区域覆盖率/数据有效性)
覆盖均一性是靶向测序最重要的参数之一,它决定了有多少目标区域能够达到分析要求。换言之,高覆盖均一性能够减少测序数据量,降低测序成本。
8#样本通过友商I** v2全外显子芯片捕获后,测序深度不足100X,故不统计其覆盖均一性。1#和4#样本测序深度相近,在10X~50X占比范围内,两款全外显子芯片均一性基本持平;由于测序深度较高,伯科v3.0芯片的其他样本均一性高于友商I** v2。
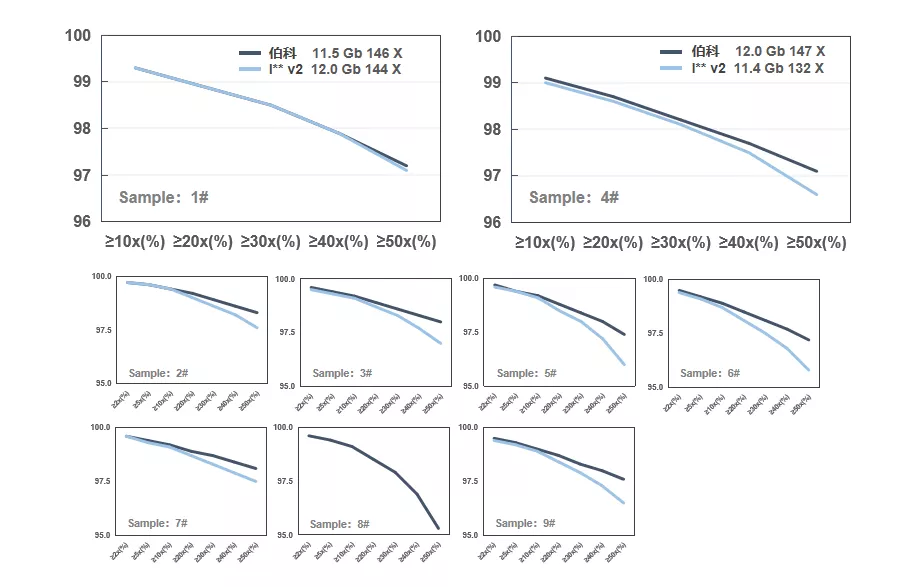
图4. 全外显子芯片捕获表现
(覆盖均一性)
综上所述,在数据比对率、数据中靶率、目标区域覆盖率方面,伯科v3.0与友商I** v2持平;在数据冗余度、数据有效性指标上,伯科v3.0略优于友商I** v2,因此,在测序相近数据量时,具有更大捕获区域的伯科v3.0仍能达到友商I** v2的测序深度(图4中1#、4#样本)。此外,两款芯片均展现出极其优异的中靶率和覆盖均一性,表明“智能映射”与“捕获感知”算法性能相近。总体而言,伯科生物TargetCap® Core Exome Panel v3.0与友商I** v2的捕获表现不相伯仲。
那么,伯科生物的外显子捕获芯片为什么会有如此优秀的覆盖均一性呢?





