大队列人群基因学研究发现克隆性造血(CH)导致癌症与心脑血管疾病
- boke
- 2024-06-12
- 2:26 下午
在过去的十年中,血液样本的基因组测序显示,克隆性造血(CH)在造血明显正常的老年人中很常见,初步估计70岁以上人群中有>10%的人患有CH。
某些特定突变的克隆肯定会导致癌症,也可以间接导致其他疾病,如心脑血管疾病。大规模回顾性研究确定了CH与血液系统恶性肿瘤、心血管疾病和全因死亡率的关系。
利用通过液相基因芯片(Gene Panel)靶向捕获技术分别检测先天遗传(冠心病多基因风险)和后天基因变异(CHIP,潜能不明克隆性造血),顾东风院士和鲁向锋教授团队首次解码了中国自然人群CHIP体细胞变异特征谱,发现CHIP体细胞变异携带会显著增加冠心病发生风险;同时,小克隆(VAF<2%)导致冠心病风险升高的作用也是不可忽视;此外,冠心病相关炎症基因可加重或抵消CHIP相关的冠心病风险,该研究于2024年1月11日发表在心脏病研究顶级期刊-JAMA Cardiology[1]。
目前,大多数CH的驱动因素仍然未知,因此需要更全面的绘制CH的驱动基因图谱,以更好地了解血液的克隆选择和衰老表型。

在最新的研究中,利用英国生物样本库(UKBB)提供的大型队列,研究人员分析了200,618名个体的血液全外显子测序(WES)数据,发现了群体水平上处于正向选择的17个新基因(ZBTB33、ZNF318、ZNF234、SPRED2、SH2B3、SRCAP、SIK3、SRSF1、CHEK2、CCD115、CCL22、BAX、YLPM1、MYD88、MTA2、MAGEC3和IGLL5),并在10,837个单细胞来源的造血细胞基因组数据中验证这种选择模式,并检测了相关的临床表型和结局[2],该研究发表在Nature Genetics期刊上。

这些基因突变的克隆在频率和大小上随着年龄的增长而增长,与经典的CH驱动因素相当,携带这些基因非同义突变(VAF>0.1)对个体健康的危害显著增加。它们与感染、死亡和血液恶性肿瘤的风险增加相关,突出了这些额外基因在衰老过程中的重要性。
全外显子测序技术可以帮助人们更全面扫描基因组,发现新的驱动基因,对于这些已经发现的驱动基因,在将来可以使用更敏感的Gene Panel检测技术、对较小的克隆扩增进行表征,进行更全面的分析。
该研究的第一作者之一Nick Bernstein指出[3]:“有了这些新发现的基因,我们现在有了更完整的理解,可以探索延缓或逆转血液中异常突变细胞过度生长的策略,以促进更健康的衰老。”。“这些基因似乎会影响炎症和免疫,这是心脏病和脑卒中等疾病的重要因素。虽然基于这项研究的干预措施还需要继续探索,但它为未来治疗各种疾病开辟了可能性。”
ApoC-II和ApoC-III在TRL代谢中的作用。LPL水解富含甘油三酯的脂蛋白(TRL)中所含的甘油三酯(TG)。脂蛋白脂酶(LPL)在脂肪细胞、骨骼肌细胞和心肌细胞中合成。LPL被LMF1激活,并通过GPIHBP1与HSPG一起转运到内皮表面。ApoC-II和ApoA-V刺激LPL活性,而ApoC-III起到抑制剂的作用[2]。
伯科全外芯片 – Core Exome Panel v3.0
TargetCap@ Core Exome Panel v3.0基于伯科高品质DNA探针合成技术开发,全流程国产制造,由~40万条探针组成,以GRCh38/hg38人类参考基因组设计,参考Refseq、CCDS、ClinVar等数据库,覆盖19,524个基因,目标区域为33.9Mb。
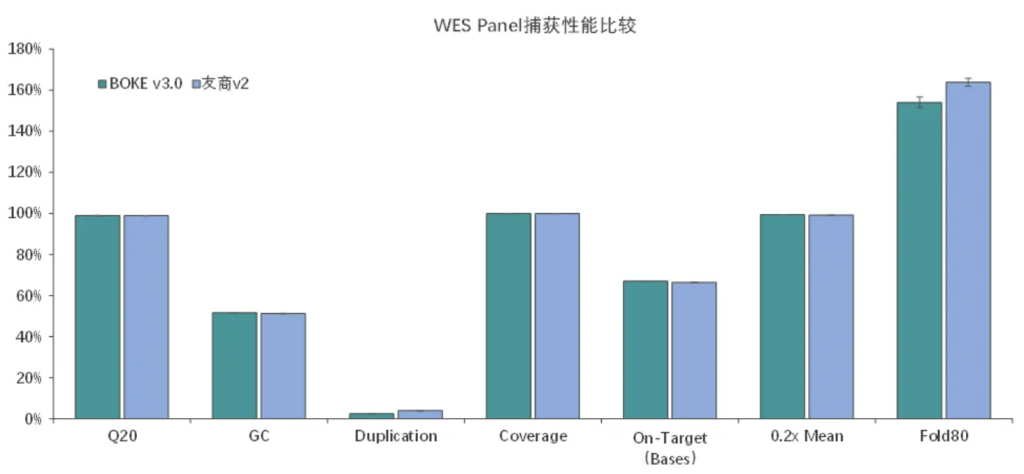
捕获性能比较
伯科全外芯片v3.0性能优异与国外友商同类型产品v2相当,中靶率、覆盖率、覆盖均一性等参数均达到国际领先水平。
适配高通量流程平台

批次稳定
使用不同批次TargetCap® Core Exome Panel v3.0芯片对NA12878 gDNA进行捕获测序,结果显示,不同批次芯片在不同测序平台上均显示出优异的稳定性,不同位点的相对深度相关性高,批次稳定。

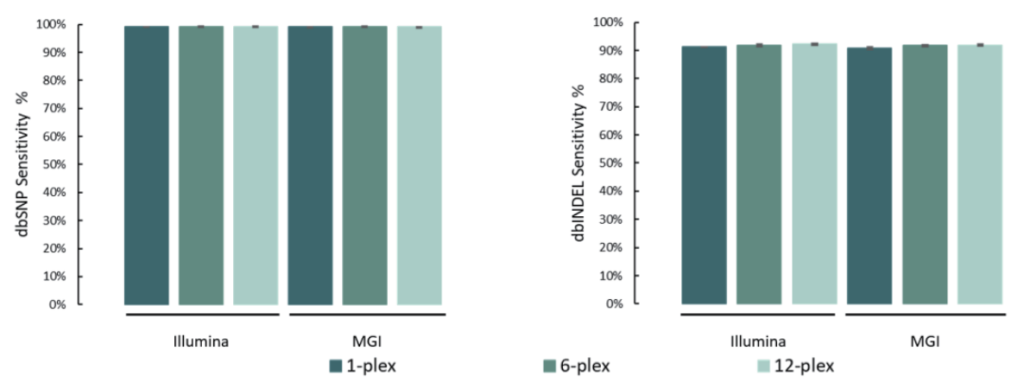
变异检测准确
单核苷酸变异(SNV)和插入缺失 (INDEL)是基因组变异的常见形式,也是引起人类疾病的重要原因。
选取NA12878标准品,与预期SNV和INDEL变异进行比较。结果表明,在MGI与Illumina测序平台,SNP灵敏度为99.1%,INDEL灵敏度为91.6%。
参考文献:
1. Zhao K, Shen X, Liu H, et al. Somatic and Germline Variants and Coronary Heart Disease in a Chinese Population[J]. JAMA cardiology, 2024.
2. Bernstein N, Spencer Chapman M, Nyamondo K, et al. Analysis of somatic mutations in whole blood from 200,618 individuals identifies pervasive positive selection and novel drivers of clonal hematopoiesis[J]. Nature Genetics, 2024: 1-9.
3. Seventeen More Genes Identified That Drive Age-Related Mutations in Blood Cells. https://www.insideprecisionmedicine.com/news-and-features/seventeen-more-genes-identified-that-drive-age-related-mutations-in-blood-cells/
4. Evans M A, Walsh K. Clonal hematopoiesis, somatic mosaicism, and age-associated disease[J]. Physiological Reviews, 2023, 103(1): 649-716.


