拨云见日 | RNA-Cap Panel 性能评估新思路,为融合基因检测保驾护航
- boke
- 2022-08-04
- 7:21 上午
基因融合事件几乎存在于所有癌症类型中,除了在癌症诊断和预后过程中扮演重要角色外,融合基因已经成为极为重要的癌症药物靶点。
恩曲替尼是继帕博利珠单抗和拉罗替尼之后,FDA批准的第三款“不限癌种”的抗癌药物,用于治疗携带NTRK基因融合的成年和青少年癌症患者,以及携带ROS1基因突变的非小细胞肺癌患者。近日,恩曲替尼(entrectinib,Roche)于2022年7月在中国获批,用于治疗NTRK融合阳性局部晚期或转移性实体瘤。目前,国内外已有多种抑制融合基因的肿瘤靶向治疗药物用于临床,包括伊马替尼/BCR-ABL1、克唑替尼/EML4-ALK和拉罗替尼/NTRK融合等。
不过,融合基因在癌症人群中的发生比例较低,因此,准确筛选出融合基因阳性患者,提高靶向用药匹配机会,将会使更多患者获益。
基于NGS的RNA靶向捕获测序(RNA-Cap)是目前公认的提高融合基因检出的技术之一,其具有以下优势:1)与传统方法相比,RNA-Cap具有高通量优势,一次性检测多个候选融合基因;2)与RNA-Seq相比,RNA-Cap对不同样本类型高度兼容,包括血液、组织和FFPE等;3)与DNA-Cap相比,RNA-Cap能有效克服基因组内含子的诸多限制和缺点,更加准确、经济。因此,RNA-Cap技术越来越多地被采用。
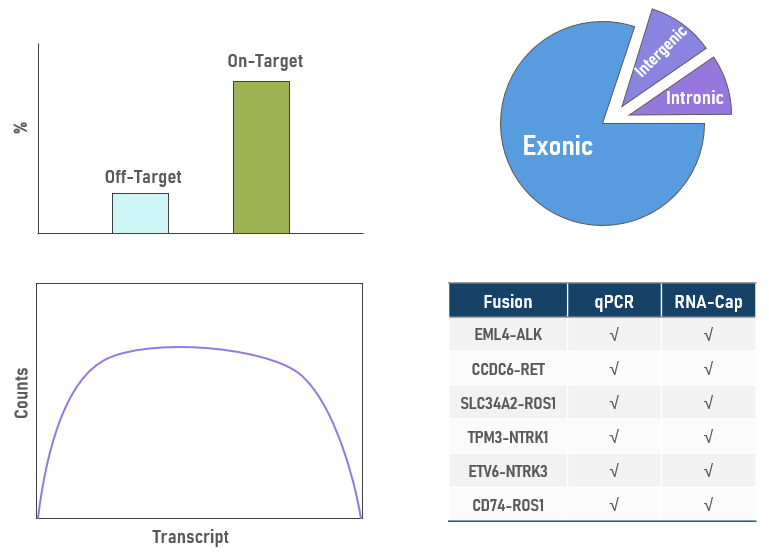
图1 常规RNA-Cap Panel性能评估
RNA-Cap Panel的性能评估通常包括目标基因中靶率、外显子数据占比、转录本覆盖均一性、融合基因检出率等(图1)。但是,由于基因表达差异和可变剪接会造成部分基因和外显子低覆盖或无覆盖(图2)。
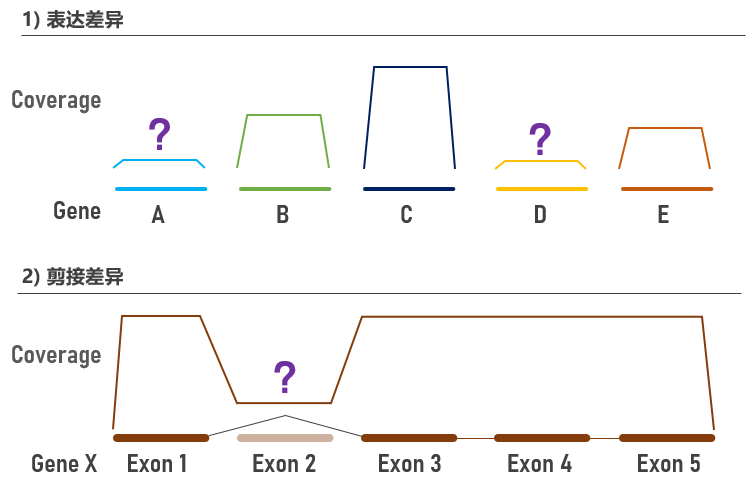
图2 RNA-Cap Panel数据中“天然”的低覆盖区域
上述问题究竟是天然的生物学现象还是探针不工作造成的呢?那么,能否寄希望于不同生物学样本的表达差异对低覆盖区域进行验证呢?我们从TCGA下载了多个细胞系的RNA-Seq数据,但不同细胞系样本的“目标”基因表达差异并不大,显然,通过不同生物学样本进行验证的路线,可行性较低。
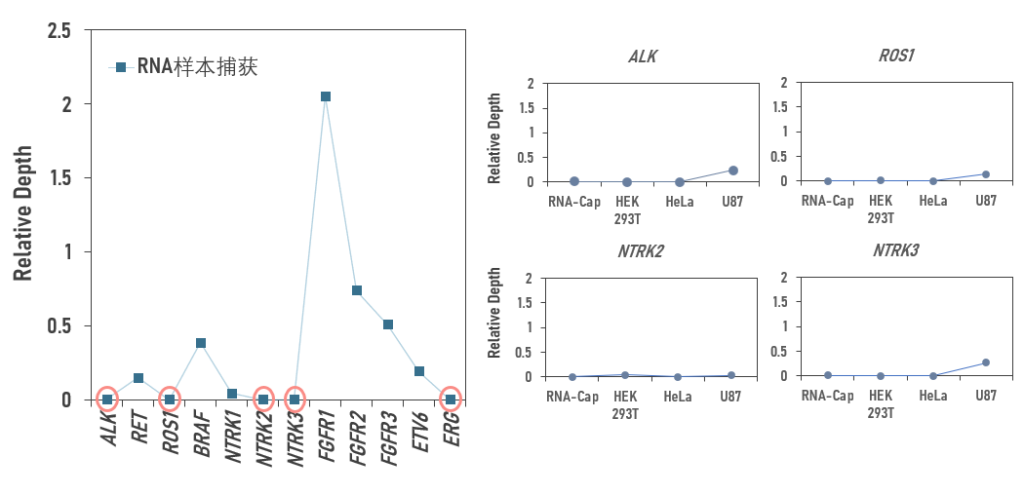
图3 RNA-Cap Panel对RNA样本进行捕获存在的低表达基因(左),低表达基因在不同细胞系中的表达量(右)
相较于转录本存在的基因表达差异,基因组层面的绝大部分基因不存在拷贝数干扰,因此,在检测RNA样本的同时,对DNA样本也平行开展RNA-Cap Panel捕获,在理论上可以解决上述问题。如图4左所示,低覆盖基因(RNA)在DNA样本中覆盖正常,相对平均深度在1附近,证明低覆盖的原因是基因低表达;同样,对于某些在RNA样本中低覆盖的外显子(图4右),在DNA样本捕获中也覆盖正常,在RNA样本中的低覆盖原因更可能来自基因的可变剪接。
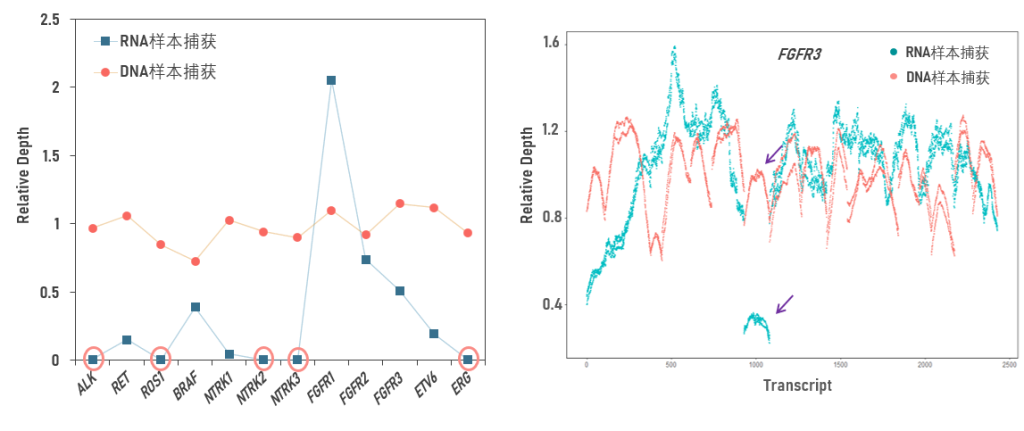
图4 RNA/DNA样本平行捕获(RNA-Cap Panel)
综上所述,在评估RNA-Cap Panel性能时,加入DNA样本捕获,采用RNA/DNA样本平行捕获评估,能够更准确的辨别基因表达和剪接差异与Panel(探针)性能的影响,特别是在不依赖阳性标准品的前提下,RNA/DNA样本平行捕获为融合热点基因或相关区域的捕获性能评估提供了新的视角。





